L'élution du gel de filtration se fait en fonction des masses molaires décroissantes. Le complexe enzyme - inhibiteur correspond au pic 1 et l'inhibiteur libre au pic 2. Donc : Pic 1 = inhibiteur lié ; pic 2 = inhibiteur libre.
| Université d'Angers - Biochimie Métabolique - Janvier 2004 |
| Voir un cours sur la représentation de Scatchard. |
| Partie
A
L'élution du gel de filtration se fait en fonction des masses molaires décroissantes. Le complexe enzyme - inhibiteur correspond au pic 1 et l'inhibiteur libre au pic 2. Donc : Pic 1 = inhibiteur lié ; pic 2 = inhibiteur libre.
|
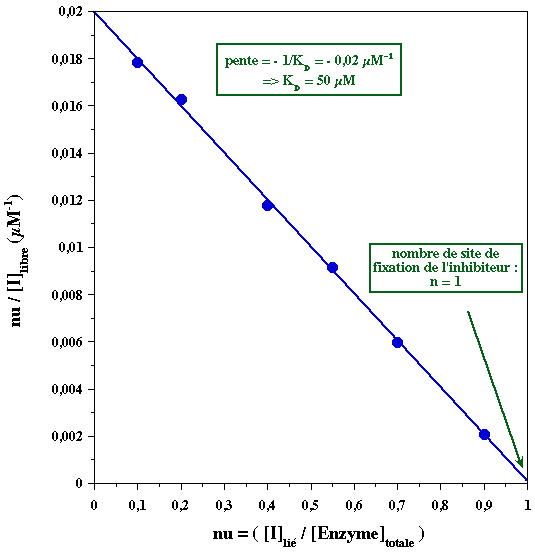
| [I]lié (µM) | [I]libre (µM) | [E]totale (µM) | nu = [I]lié / [E]tot | nu / [I]libre (µM-1) |
| 10 | 5.60 | 100 | 0.100 | 0.0179 |
| 20 | 12.3 | 100 | 0.200 | 0.0163 |
| 40 | 34.0 | 100 | 0.400 | 0.0118 |
| 55 | 60.0 | 100 | 0.550 | 0.00917 |
| 70 | 117 | 100 | 0.700 | 0.00598 |
| 90 | 437 | 100 | 0.900 | 0.00206 |
Remarque : 1 molécule d'inhibiteur se fixe par molécule d'enzyme. Masse molaire de l'enzyme = 25 kDa et masse molaire du complexe = 75 kDa => masse molaire de l'inhibiteur = 50 kDa Une stoechiométrie 1:1 est donc cohérente avec la masse molaire du complexe et avec l'ordre d'élution complexe puis inhibiteur (l'enzyme libre étant exclue du gel). |
|
Partie B 1. En absence d'inhibiteur : VMax = 1,18 UA.min-1
D'après la relation de Beer - Lambert et avec εMproduit = 2600 M-1.cm-1 et l = 1 cm : 1,18
VMax 2. En présence de l'inhibiteur : VMaxapp = 1,9 10-4 M.min-1 = 3,2 µM.sec-1 VMaxapp < VMax et KMS est inchangé => inhibition non compétitive. VMaxapp . [I]0
|