a. Caractéristiques générales
Le phage λ gt11 possède un ADN double brin encapsidé dans une tête icosahèdrale.
Il possède une mutation "ambre" (sam100 ou S100) dans les gènes codant pour les protéines de la capside (gènes de la lyse). Ceci limite les risques de propagation de bactériophages recombinants dans des souches non transfectées conservées au laboratoire.
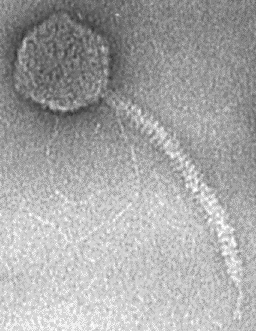
Source : Maria Schnos - University of Wisconsin - Madison
- Le phage λ gt11 code aussi pour un répresseur sensible à la température (cIts857).
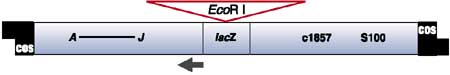
Par ailleurs certaines souches E. coli hôte possède un gène codant pour un suppresseur de mutation "ambre". C'est le cas de LE 392 qui possède le gène supF qui code pour un suppresseur ARNt qui introduit une tyrosine au niveau du codon UAG.
Cette souche est donc appropriée à la formation de plages de lyse abondantes (à une température où le répresseur est actif) et est utilisée pour la détermination du titre de la banque (voir la partie lysogénie & lyse).