| Quelques méthodes d'étude des interactions protéine - protéine |
| Voir le cours sur les méthodes d'étude des interactions protéine - protéine. |
|
Exercice N°1 1. Associer chaque puissance de 10 à un symbole du Système International d'Unités
|
2. Ordre de grandeur de différents phénomènes physiques concernant les macromolécules biologiques
Source : Kalyaanamoorthy & Chen (2014) Excitation, émission de fluorescence, ... : voir une figure et un tableau de valeurs. Quelques valeurs extraites de la littérature :
|
| Liaison - Longueur | Diamètre |
| C-H : 1,07 Å | atome # 10-10 m (1 Å) |
| N-H : 1,01 Å | noyau # 10-15 m (1 fm) |
| O-H : 0,96 Å | ------ |
Exercice N°2 1. Convertir 1 µm3 en pL. Convertir 1 pL en µm-6.
2. Soit une cellule d'un volume de 103 µm3 avec une concentration totale de protéines = 5 µM :
|
a. Concentration totale de protéines : 5 µM (5 10-6 moles.L-1) dans 1 cellule de volume 1 pL Soit : 5 10-6 moles dans 1012 pL => 5 10-18 moles dans 1 pL |
x = (5 10-18 . 6,022 1023 / 1) ≈ 3 106 molécules de protéines dans 1 cellule de volume 1 pL Et : (3 106 / 104) ≈ 300 molécules de protéines d'un type donné |
b. Nombre d'Avogadro (N) : 1 mole -> 6,022 1023 molécules soit : 5 10-18 moles -> x molécules |
| 3. Aller au site "B10NUMB3R5" : choisir des mots-clés adaptés pour obtenir des valeurs du nombre de molécules protéines dans une cellule. |
Exercice N°3 1. Quel est le nom complet des méthodes d'études des interactions protéine-protéine dont les abréviations suivent : CG-MALS; AUC; SEC-MALS; BLI; CE; SPR; ITC ?
Quels sont leurs points communs et leur intérêt ? Ce sont des méthodes optiques qui ne nécessitent aucun marquage (sans étiquette) : travail simplifié et aucune perturbation induites par le marquage. |
Elles sont classées en 3 catégories : basées sur la surface, basées sur la séparation et basées sur une solution.
Source : Soltermann et al. (2021) |
2. Illustrer 2 processus cellulaires contrôlés par des interactions protéine-protéine (en mentionnant quelques exemples) et se déroulant dans des organites distincts.
Illustration : les protéines multilocalisées jouent un rôle important dans les réseaux d'interactions protéine-protéine chez Arabidopsis thaliana.
|
Exercice N°4 Interpréter l'enregistrement ci-dessous (nom de la technique, différentes phases, données obtenues, …). Il s'agit d'un sensorgramme de résonance de plasmons de surface ("Surface Plasmon Resonance" - SPR) : c'est une méthode optique qui mesure l'indice de réfraction à la surface d'un biocapteur. Une protéine est immobilisée sur une surface métallique (le biocapteur) et une solution du ligand est injectée sur cette surface.
|
Exercice N°5 Interpréter la figure ci-dessous (nom de la technique, différentes phases, données obtenues, …).
Source : Wrapp et al. (2020) Les protéines de pointe S ("spike") virales 2019-nCoV S et SARS-CoV S ont le même récepteur fonctionnel de la cellule hôte, l'enzyme de conversion de l'angiotensine 2 (ACE2).
Résultats
Interprétation
|
Exercice N°6 : Interférométrie de biocouche ("Bio-layer interferometry" - BLI) appliquée à l'interaction crRNA - CRISPR-Cas Cette technique mesure le décalage de longueur d'onde de réflection de la lumière blanche ("Reflected beam") en fonction de la variation d'épaisseur d'une biocouche (par rapport à une couche de référence - "Optical biolayer"). La variation d'épaisseur de la biocouche dépend elle-même de la concentration du complexe biologique formé, donc des concentrations des molécules qui interagissent et de leur constante de dissociation KD.
Source : Müller-Esparza et al. (2020) Par ailleurs, les complexes ribonucléoprotéiques sont formés par un ARN guide CRISPR (appelé crRNA) et des protéines associées à CRISPR ("CRISPR-associated" - Cas). Voir un rappel sur les types de mécanismes CRISPR-Cas (types I à VI et 33 sous-types). |
Dans cet exemple, la biocouche ("Biosensor") est recouverte de streptavidine 100 nM ("Streptavidine biolayer" - phase d'enregistrement "Baseline") qui fixe des oligonucléotides marqués par la biotine en 5' (chaînes en vert - phase d'enregistrement "Oligo binding"). Ces longs oligonucléotides d'ADN :
Source : Müller-Esparza et al. (2020) Phases de l'enregistrement du signal
|
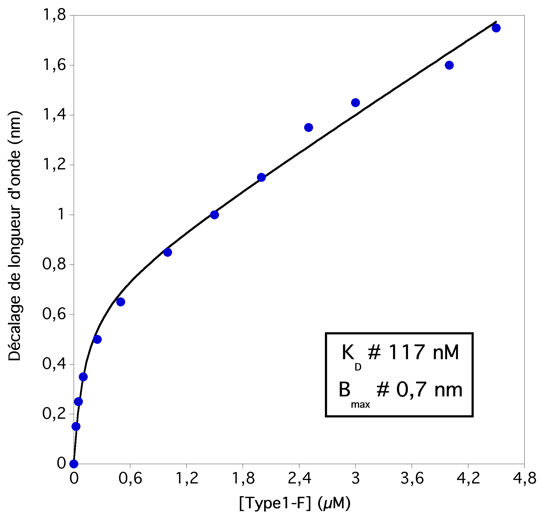
Résultat avec le complexe effecteur de type 1-Fv deShewanella putrefaciens
|
| [Type I-Fv] (µM) | Décalage λ (nm) | [Type I-Fv] (µM) | Décalage λ (nm) |
| 0 | 0 | 1,5 | 1 |
| 0,025 | 0,15 | 2 | 1,15 |
| 0,05 | 0,25 | 2,5 | 1,35 |
| 0,1 | 0,35 | 3 | 1,45 |
| 0,25 | 0,5 | 4 | 1,6 |
| 0,5 | 0,65 | 4,5 | 1,75 |
| 1 | 0,85 |
Résultats
|
 |
Exercice N°7 - Titrage calorimétrique isotherme (ITC) Analyser les graphiques ci-dessous. Recalculer les valeurs indiquées sur ces figures.
Source : The ITC experiment |
| Questions | Réponses |
| Q1. Quels types de force sont impliqués dans les interactions protéine-protéine ? | Les liaisons de Van der Waals, liaisons ioniques, interactions hydrophobes, ... |
| Q2. Quel(s) rôle(s) ont les molécules d'eau dans les interactions protéine-protéine ? | La dynamique conformationnelle et la concentration des ions et molécules. Voir les rôles de la molécule d'eau dans les interactions entre ions ou molécules. |
| Q3. Quelles sont les 2 conditions pour qu'il y ait une interaction protéine-protéine ? | La rencontre (localisation, concentration et vitesse des molécules) La complémentarité des structures |
| Q4. Quelle grandeur thermodynamique est directement mesurée par la technique ITC ? | La variation d'enthalpie (ΔH) |
| Q5. Comment s'appelle une protéine d'intérêt liée covalamment (synthèse peptidique) à une GFP ? | Une protéine de fusion |
| Q6. Citer 3 critères de choix d'une technique pour étudier les interactions protéine-protéine. | Méthodes sans marquage ou sans immobilisation. Possibilité d'étudier une large gamme de concentrations du ligand (détermination d'une large gamme de valeurs de KD). Possibilité de mesurer les valeurs de ka et kd. La quantité disponible de molécule(s) étudiée(s). |
| Q7. Comment s'appelle la classe de molécules synthétiques qui accroissent drastiquement la fixation d'une protéine sur une autre ? | Inducteur chimique de proximité (ICP) |
| Q8. Comment s'appelle un modèle d'apprentissage profond qui s'appuie sur un corpus de connaissances textuelles colossal pour s'entraîner ? | Un modèle de langage pré-entraînés pour le traitement du langage naturel ("large natural language models"). |
| Q9. Hormis la fluorescence, sur quel principe identique s'appuient les techniques double-hybrides et fluorescence par complémentation bimoléculaire ? | La complémentarité de structure de 2 protéines qui reconstituent une protéine fonctionnelle. |
| Q10. Citer 2 qualités de la techniques BLI. | Insensible aux molécules de ligand non liées, aux variations d'indice de réfraction ou de débit du milieu circulant. Etude d'une large gamme de valeurs de KD. |
| Assertions | Réponses |
| A1. Seules les protéines ont une dynamique conformationnelle. | FAUX |
| A2. Toutes les méthodes d'étude des interactions protéine-protéine permettent de déterminer la cinétique d'association de ces protéines. | FAUX |
| A3. Toutes les méthodes d'étude des interactions protéine-protéine permettent de déterminer la constante de dissociation de ces protéines. | VRAI |
| A4. Une technique utilisant une/des protéine(s) de fusion est rapide et simple à appliquer. | FAUX |
| A5. Les anticorps sont un outils important pour l'étude des interactions protéine-protéine. | VRAI |
| A6. La technique appelée double-hybrides est une technique à très haut débit. | VRAI |
| A7. La BLI et la SPR génèrent des enregistrements d'allures sensiblement identiques. | VRAI |
| A8. La réponse mesurée en SPR peut traduire un décalage d'angle critique de plusieurs degrés. | FAUX |
| A9. Plus le nombre de molécules d'un ligand se fixant sur les sites de fixation d'une protéine est élevé, plus la variation d'enthalpie de cette réaction est faible. | FAUX |
| A10. Plus KD est petite, plus l'affinité de liaison du ligand pour son site de fixation est grande. | VRAI |
| Retour à l'ensemble de ressources sur les interactions entre biomolécules. |