
| La méthylation |
| Tweet |
 |
|
1. Introduction 2. Protéine méthyltransferases et S-adénosyl-L-méthionine 3. La protéine L-isoaspartate O-méthyltransferase |
4. Rôles de la méthylation de l'arginine 5. Régulation de la transcription par la coactivateur-associé arginine méthyltransferase 1 (CARM1) 6. Liens Internet et références bibliographiques |
|
1. Introduction La méthylation consiste à ajouter un ou plusieurs groupement(s) méthyle (-CH3). Exemples de protéines méthylées : la calmoduline, la myosine, l'actine, la calcineurine, la rhodopsine, le cytochrome c. Les acides aminés méthylés sont : l'alanine, la lysine, la methionine, l'histidine, l'asparagine, l'acide aspartique ou glutamique, l'arginine et les groupes NH2 et COOH terminaux. La méthylation est impliquée dans des processus biologiques tels que les interactions protéine - protéine, la localisation cellulaire, la transduction du signal, la régulation dite épigénétique de la transcription des gènes via la modification des histones. |
| Acide aminé modifié | Enzyme de la modification | Structure de l'acide aminé modifié | Exemples de protéines modifiées |
| N,N,N-trimethyl-L-alanine | protéine ribosomale L11 méthyltransférase prmA |
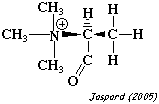
|
|
| N-methyl-L-méthionine | S-adénosylméthionine-methionyl-peptide N-méthyltransférase |
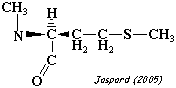
|
|
| N4-methyl-L-asparagine | phycobiliprotéine asparagine N-méthyltransférase |
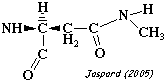
|
|
| N5-methyl-L-arginine | protéine-arginine N5-méthyltransférase |
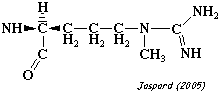
|
La méthylation des arginines des histones joue un rôle important dans la régulation épigénétique de la transcription des gènes. |
| 2. Protéine méthyltransferases et S-adénosyl-L-méthionine La méthylation des acides aminés après leur incorporation dans la chaîne polypeptidique au cours de la traduction est catalysée par le groupe d'enzymes appelées protéine méthyltransferases (à ne pas confondre avec la protéine O-méthyltransferase qui est une enzyme de ce groupe). Le donneur activé de groupement méthyle est la S-adénosyl-L-méthionine qui est synthétisée par la S-adénosylméthionine synthétase ou methionine adénosyltransferase (EC 2.5.1.6) : ATP + L-méthionine + H2O <=> phosphate + diphosphate + S-adénosyl-L-méthionine
|
|
3. La protéine L-isoaspartate O-méthyltransferase La réaction catalysée par la protéine-L-isoaspartate(D-aspartate) O-méthyltransferase (EC 2.1.1.77) est : S-adénosyl-L-méthionine + protéine L-isoaspartate <=> S-adénosyl-L-homocysteine + protéine L-isoaspartate α-méthyl ester
Source : KEGG |
4. Rôles de la méthylation de l'arginine De très nombreuses protéines dont les arginines sont méthylées sont impliquées dans des processus associés au métabolisme des ARN (biogenèse, traitement, épissage et expression des gènes), dans l'organisation de la chromatine, la traduction et les processus métaboliques et microtubulaires. En conséquence, ces protéines sont localisées dans le noyau, le nucléole, les ribosomes, les granules de ribonucléoprotéine (RNP), les microtubules ... La méthylation de l'arginine (acide aminé le plus basique) modifie le groupe guanidinium qui est protoné au pH physiologique. Or cette charge positive médie de nombreuses interactions intramoléculaires et intermoléculaires (y compris liaison hydrogène et empilement).
La méthylation de l'arginine affaiblit également l'association des protéines riche en arginine avec les acides nucléiques. En particulier, les séquences RG ou RGG ont tendance à se lier à l'ARN :
Les séquences RG, RGG et GRG sont présentes (jusqu'à 20 répétitions) dans de nombreuses protéines. Les motifs RG se trouvent dans des régions de faible complexité structurale ou des régions intrinsèquement désordonnées qui ont une propension à former des bio-condensats. La formation de bio-condensats est un type d'organisation dans l'assemblage et la fonction des organites sans membrane tels que le nucléole, les granules de stress et les corps P : ces organites contiennent des protéines et de l'ARN et régulent la transcription, la traduction et la réponse au stress. |
Régulation métabolique de la méthylation de l'arginine La méthylation de l'arginine par les protéines arginine méthyltransférases (PRMT) nécessite la S-adénosyl-L-méthionine (SAM) : en effet, SAM est le cofacteur donneur de méthyle des PRMT pour méthyler le groupe guanidinium de l'arginine. De nombreuses PRMT (PRMT1 à PRMT9) effectuent cette réaction de méthylation (CARM1 est PRMT4).
Source : Wu et al. (2021) Inhibitions
La monométhylarginine circulante (MMA) et l'arginine diméthylée asymétrique (ADMA) peuvent inhiber la fonction de l'oxyde nitrique synthase (NoS), l'enzyme qui génère l'oxyde nitrique (No), un puissant vasodilatateur qui régule la pression artérielle et l'inflammation : |
5. Régulation de la transcription par la coactivateur-associé arginine méthyltransférase 1 (CARM1) CARM1 est homologue des protéines arginine méthyltransférases. C'est une protéine de 608 acides aminés chez l'homme et la souris. Elle est associée aux coactivateurs de transcription de la classe p160 impliqués dans l'activation des gènes. CARM1 interagit aussi avec les coactivateurs de transcription CBP/p300. CARM1 méthyle l'arginine 2142 (domaine de fixation de GRIP1) de p300. L'une des cibles de CARM1 sont les histones H3 (arginine 17) et H4, qui sont aussi cibles de l'activité acétylase des coactivateurs CBP/p300 : en se fixant aux régions des promoteurs via les coactivateurs, CARM1 augmente la méthylation des histones et rend ces régions plus accessibles pour la transcription.
Source : Biocarta |
| 6. Liens Internet et références bibliographiques |
|
MeMo: Methylation Modification Prediction Server (Arginine and Lysine sites) RESID Database of Protein Modifications |
|
|
Billingsley et al. (1985) "Stoichiometric methylation of calcineurin by protein carboxyl O-methyltransferase and its effects on calmodulin-stimulated phosphatase activity" PNAS 82, 5612 - 5616 Ananthanarayanan et al. (2004) "Ligand-dependent Activation of the Farnesoid X-receptor Directs Arginine Methylation of Histone H3 by CARM1" J. Biol. Chem. 279, 54348 - 54357 Wu et al. (2021) "Protein arginine methylation: from enigmatic functions to therapeutic targeting" Nat. Rev. Drug Discov. 20, 509 - 530 |