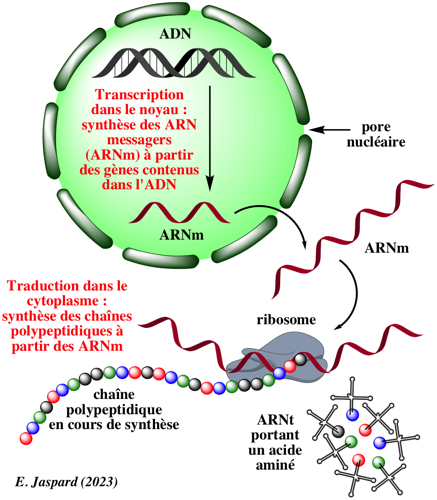
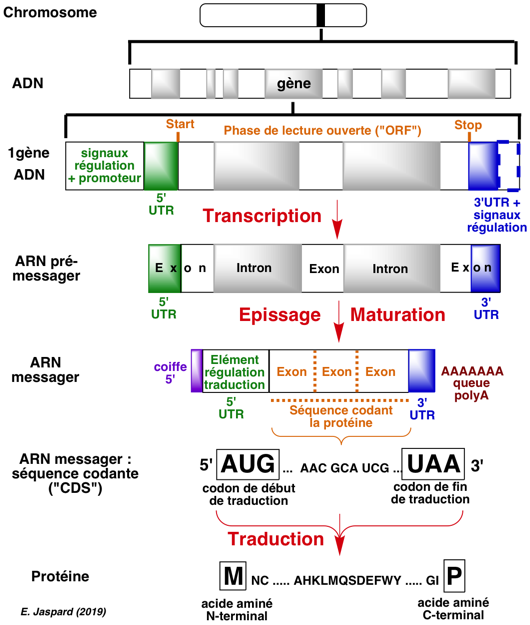
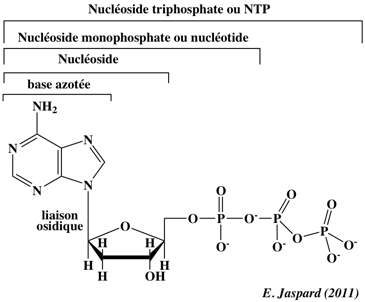
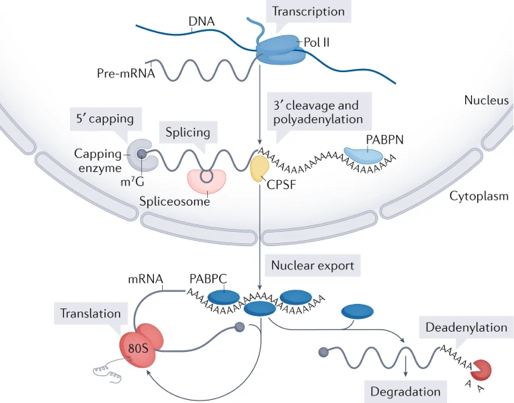
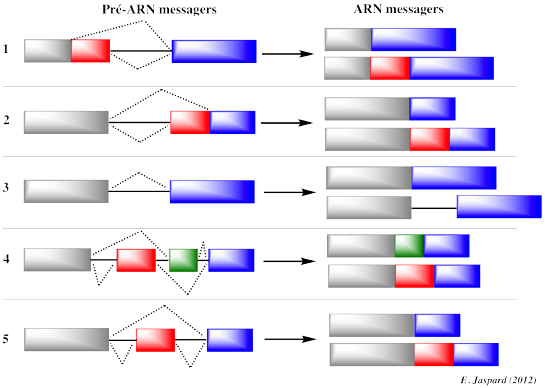
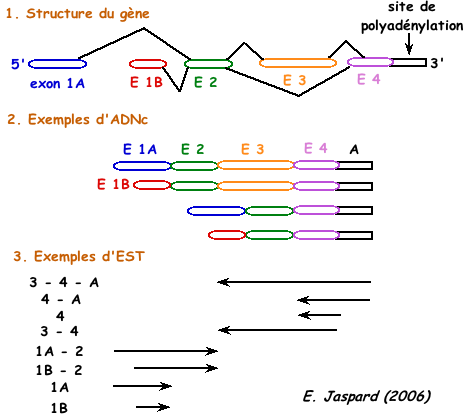
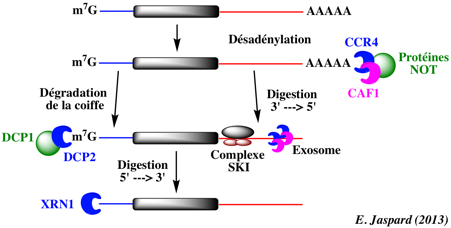
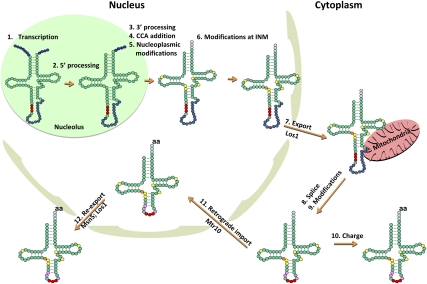
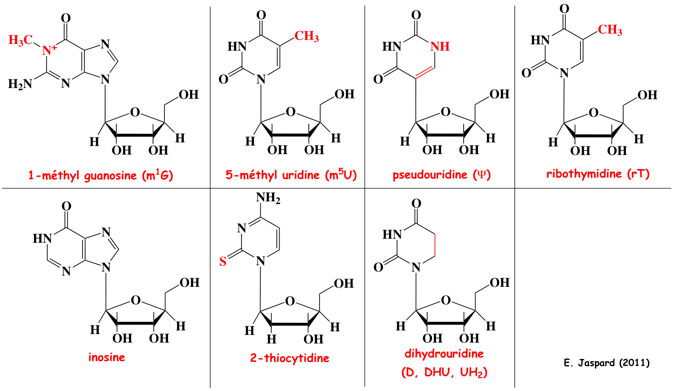
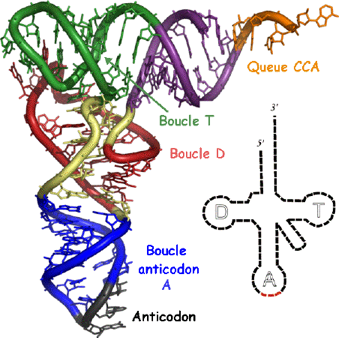
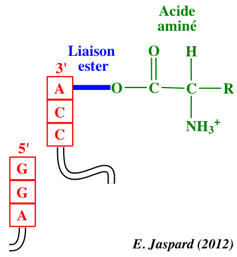
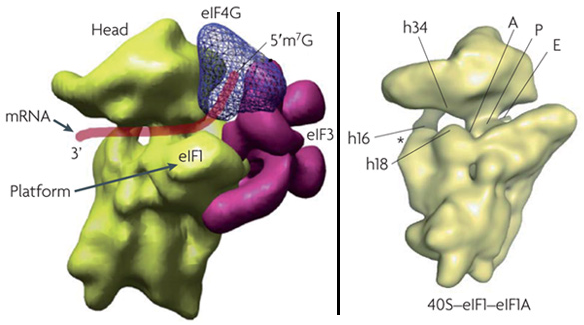
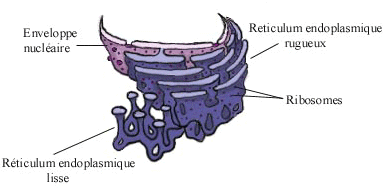
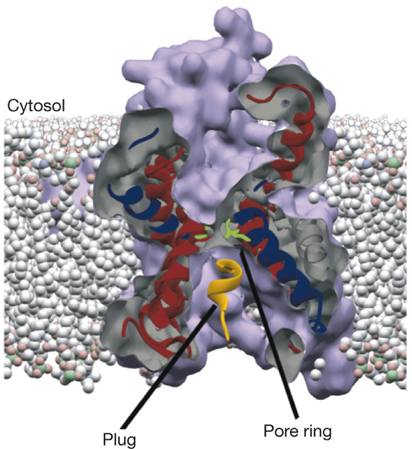
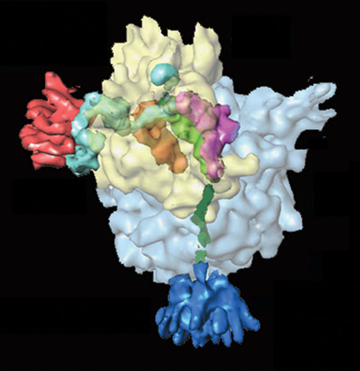
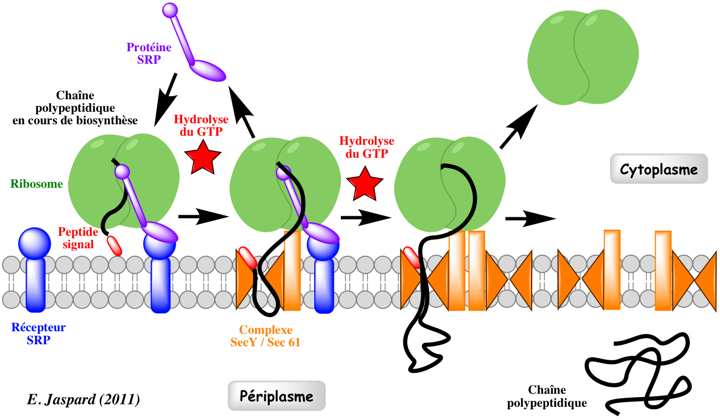
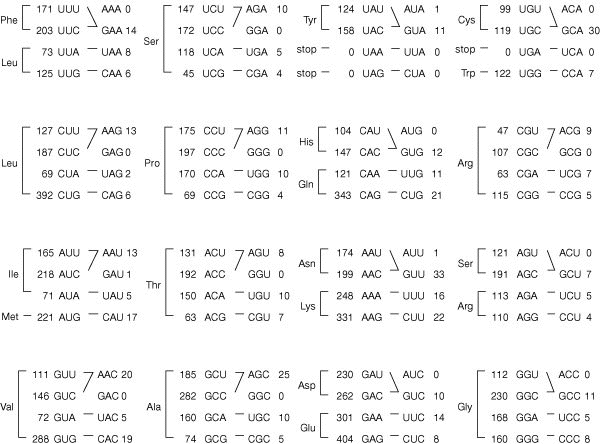|
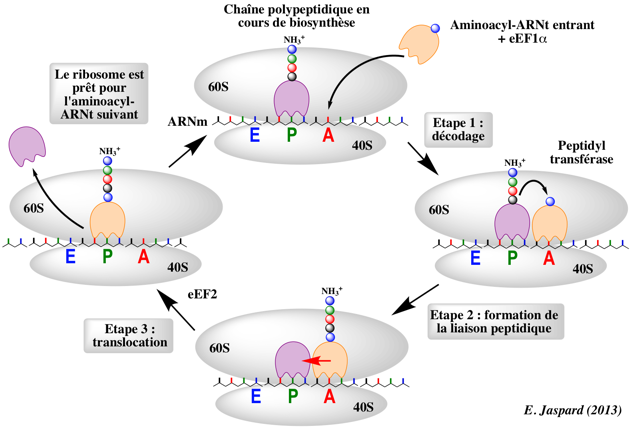
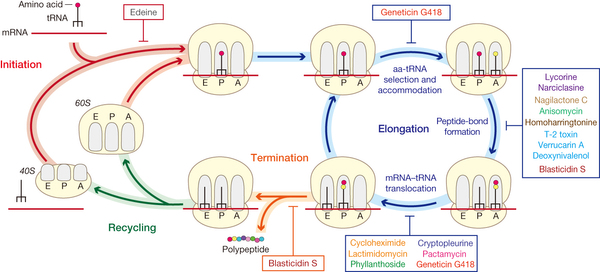
b. Elongation de la traduction
L'élongation nécessitent des facteurs d'élongation :
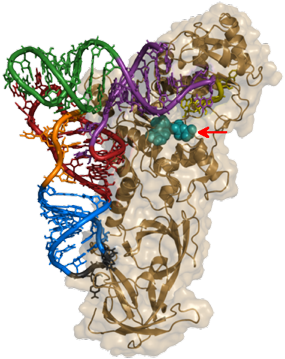
- EF-Tu (appelé aussi EF1A ou EF1-α) : responsable de la sélection et de la liaison de l'aminoacyl-ARNt apparenté au site A (site accepteur) du ribosome.
- EF-Ts (ou EF1B ou EF1-β/γ/δ) : facteur d'échange de nucléotides qui régénère EF1A de sa forme inactive (EF1A-GDP) en sa forme active (EF1A-GTP). EF-Ts est plus complexe chez les eucaryotes que chez les bactéries.
- EF2 (ou EF-G) : responsable de la translocation du peptidyl-ARNt du site A au site P (site peptidyl-ARNt), libérant ainsi le site A pour l'aminoacyl-ARNt suivant.
EF-Tu et EF2 sont des petites protéines fixant le GTP ("small GTP-binding proteins").
Etape 1 : décodage
EF-Tu-GTP se fixe et délivre un aminoacyl-ARNt au site A du ribosome. EF-Tu reconnaît et fixe tous les aminoacyl-ARNt avec à peu près la même affinité (quand chaque ARNt est lié à l'acide aminé correct).
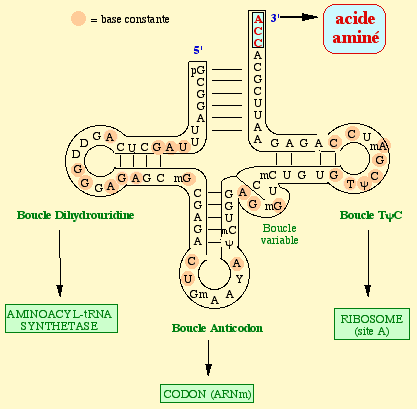
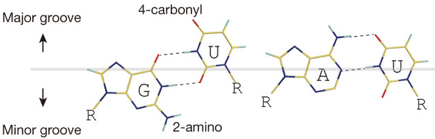
L'ARNt doit avoir l'anticodon correct pour interagir avec le codon de l'ARNm positionné sur le site A pour établir une paire de bases avec la bonne géométrie. Les bases universellement conservées de l'ARNr 16S (chez les procaryotes) interagissent avec et imprime la configuration du petit sillon ("minor groove") de la courte portion de double hélice formée par les 2 premières paires de bases du complexe [codon/anticodon].
Une conformation particulière du ribosome est stabilisée par cette interaction, ce qui fournit un mécanisme pour détecter si l'ARNt lié est correct. La relecture - correction (activité "proofreading") implique en partie la libération de l'aminoacyl-ARNt avant la formation de la liaison peptidique, si la conformation du ribosome générée par cette interaction n'est pas correcte.
Le changement de conformation du ribosome qui résulte de la formation du complexe [codon-anticodon] correct entraîne un changement de conformation du site actif de EF-Tu fixé ce qui induit l'apparition de son activité GTPase.
Quand EF-Tu délivre l'aminoacyl-ARNt au ribosome, l'ARNt possède initialement une conformation déformée. Quand le GTP fixé sur EF-Tu est hydrolysé en [GDP + Pi], EF-Tu subit un grand changement de conformation et se dissocie du complexe. La conformation de l'ARNt se relâche et le bras accepteur est repositionnée pour favoriser la formation de la liaison peptidique. Ce processus s'appelle l'accomodation.
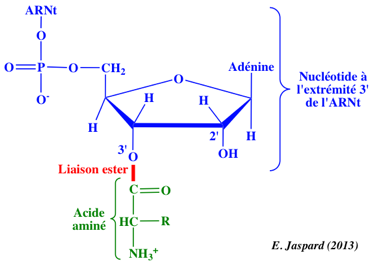
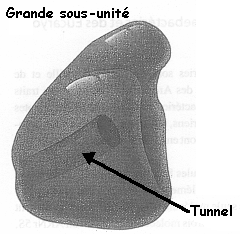
L'accomodation inclue la rotation de l'extrémité 3' simple brin du bras accepteur de l'ARNt au site A autour d'un axe qui traverse le centre peptidyl transférase de la grande sous-unité du ribosome. Ceci positionne l'extrémité 3' avec son acide aminé attaché dans le site actif près de l'extrémité 3' de l'ARNt au site P, et près de l'embouchure du tunnel par lequel le polypeptide naissant quitte le ribosome.
Voir un site extrêmement détaillé sur ce processus.
L'interaction de EF-Ts avec EF-Tu libère le GDP de EF-Tu. Après dissociation de EF-Ts, EF-Tu fixe de nouveau le GTP présent dans le cytosol à une concentration plus élevée que le GDP.
Etape 2 : formation de la liaison peptidique
La transpeptidation ou formation de la liaison peptidique implique une attaque nucléophile du groupement aminé de l'acide aminé lié au groupement hydroxyle en 3' de l'adénosine terminale de l'ARNt au site A sur le groupement carbonyle de l'acide aminé (auquel est attaché le polypeptide naissant) lié par une liaison ester à l'ARNt au site P.
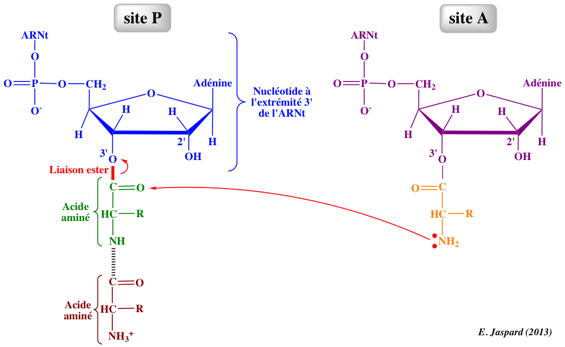
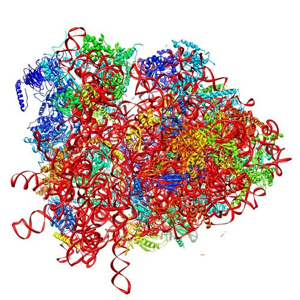
La formation de la liaison peptidique est catalysée par le ribosome lui-même : elle est favorisée par la géométrie du "site actif" de l'ARNr 23S (chez les procaryotes) de la grande sous-unité ribosomique.
L'ARNr 23S peut être considéré comme un ribozyme à activité peptidyl transférase.
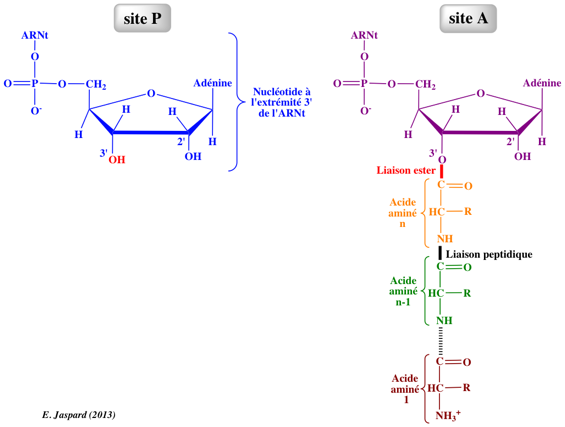
Etape 3 : translocation
Le ribosome déplace un codon vers l'avant sur l'ARNm. Le codon suivant de l'ARNm est disponible pour l'interaction avec un nouvel aminaoacyl-ARNt au site A.
Simultanément, l'ARNt "vide" est décalé du site P vers le site E et le peptidyl-ARNt est transloqué du site A au site P. Le processus est facilité par le facteur d'élongation EF2 et le GTP.
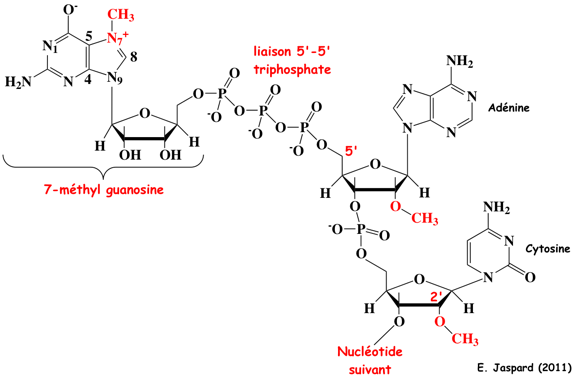
La wybutosine :
- C'est un analogue de nucléoside hypermodifié (dérivé de la guanosine) constitué de la base wyosine avec un groupe 4-méthoxy-3-[(méthoxycarbonyl)amino]-4-oxobutyle.
- La wybutosine se situe en position 37 de l'ARN de transfert Phe (ARNtPhe) des eucaryotes et améliore la précision de la traduction.
- La wybutosine s'empile sur la première paire de bases [codon-anticodon] formée par [le codon UUC de l'ARNm et l'ARNtPhe dans l'état ap/P] : la wybutosine stabilise la dernière position du codon AUG adjacent en amont et, en stabilisant de la sorte l'ARNm, empêche un décalage du cadre lors de la translocation.
c. Terminaison de la traduction
Ces étapes sont répétées jusqu'à ce que le ribosome rencontre un codon STOP dans le cadre de lecture: UAG, UAA ou UGA. La traduction se termine quand l'un de ces codons occupe le site A.
Les codons STOP n'ont aucun ARNt correspondant. Au lieu de cela, les codons STOP sont reconnus par la protéine RF1 ("Releasing Factor 1" - facteur de libération 1) ou eRF chez les eucaryotes (il y a 2 facteurs de terminaison chez les procaryotes et un seul chez les eucaryotes). L'activité de RF1 est stimulée par RF3 (une protéine fixant le GTP).
Le complexe [RF1 / RF3] se lie aux ribosomes qui ont un codon STOP au site A : il stimule le clivage de la liaison ester entre le peptide et le peptidyl-ARNt, libérant ainsi la nouvelle protéine du ribosome. Les facteurs sont libérés du ribosome après hydrolyse du GTP.
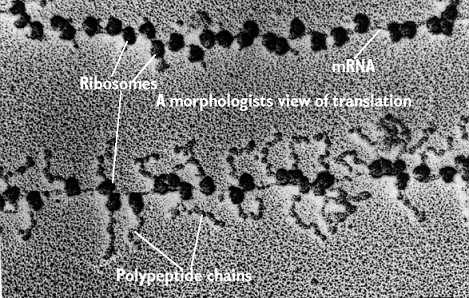
Le ribosome commence un nouveau cycle de traduction sur le même ARNm dans la plupart des cas. |