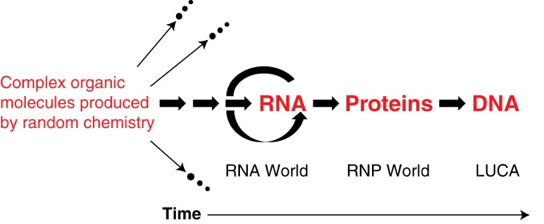
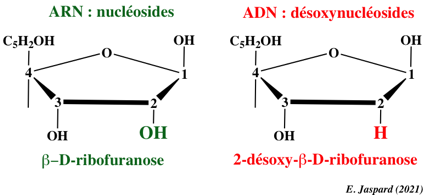
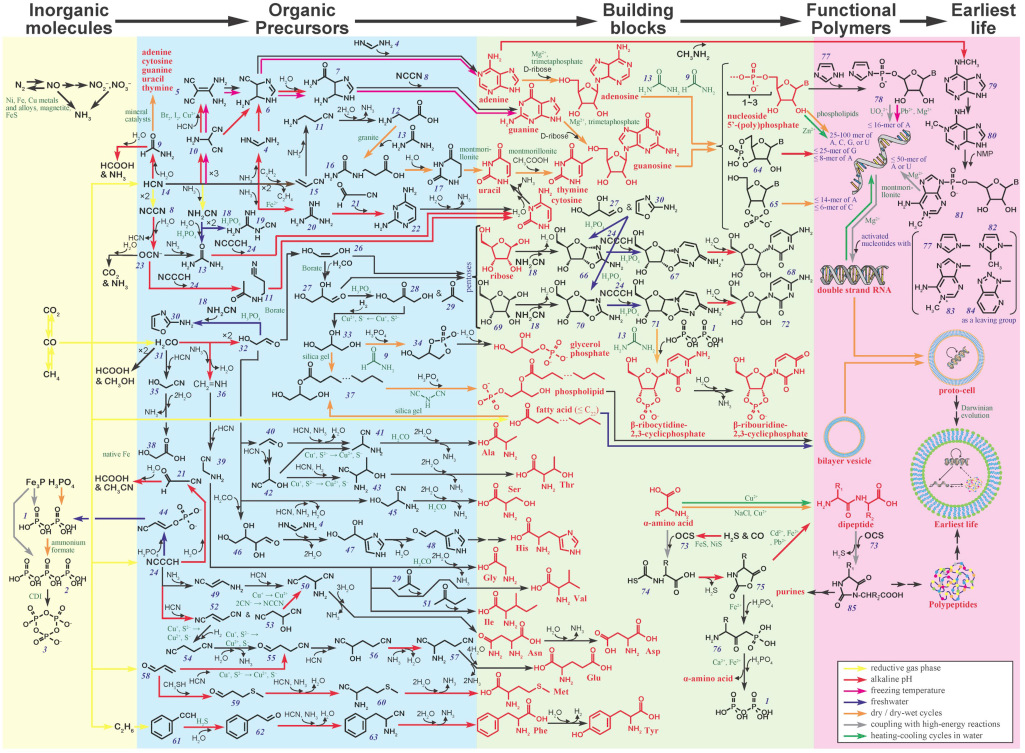
3. Les ribonucléotides et l'acide ribonucléique (ARN)
a. Structure générale
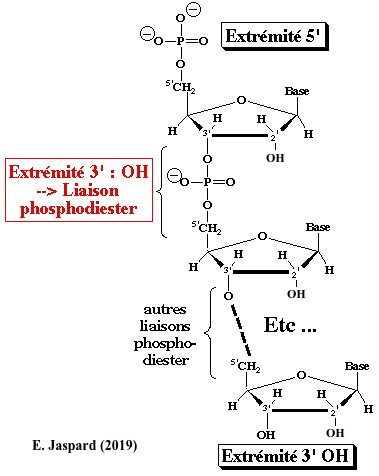
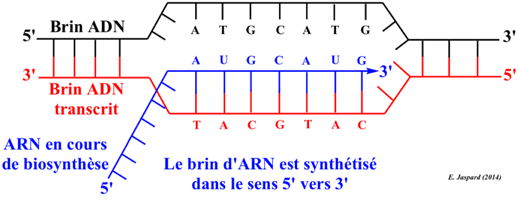
La molécule d'ARN est un polymère linéaire composé de 4 nucléotides : l'adénosine monophosphate (AMP), l'uridine monophosphate (UMP), la guanosine monophosphate (GMP) et la cytidine monophosphate (CMP). Chaque nucléotide est constitué d'une base aromatique plane attachée à un ribose (cycle furanose) et d'un groupe 5'-phosphate.
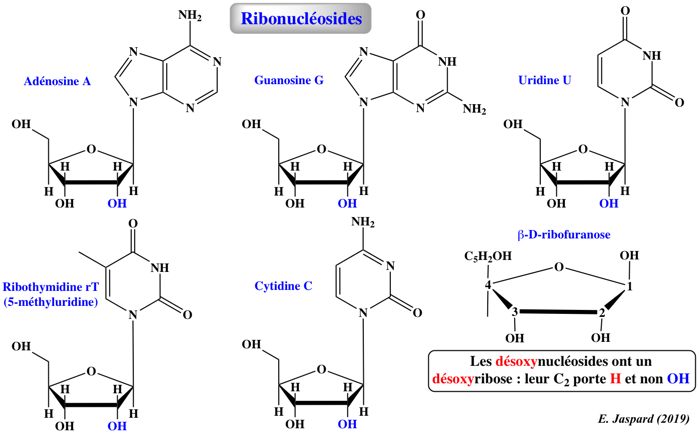
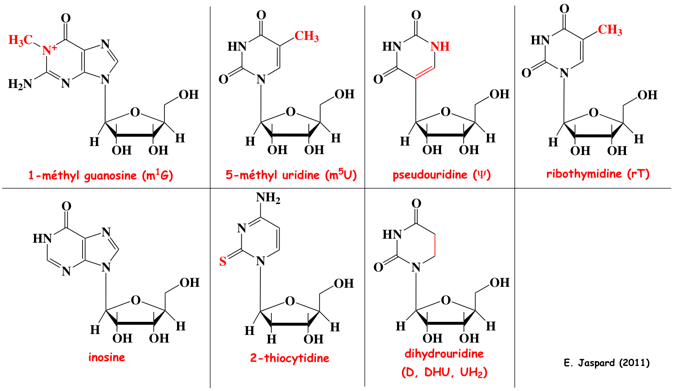
La ribothymidine est trouvée essentiellement dans la boucle T des ARN de transfert (ARNt) à laquelle elle donne son nom. Lors de la transcription, une uridine est incorporée en position 54 dans le précurseur de l'ARNt puis cette uridine est modifiée par l'ARNt (uracile(54)-C(5))-méthyltransférase (EC 2.1.1.35) en ribothymidine. Elle joue un role important dans la stabilisation de la structure 3D des ARNt.
Conséquence du groupe hydroxyle 2'OH du ribose
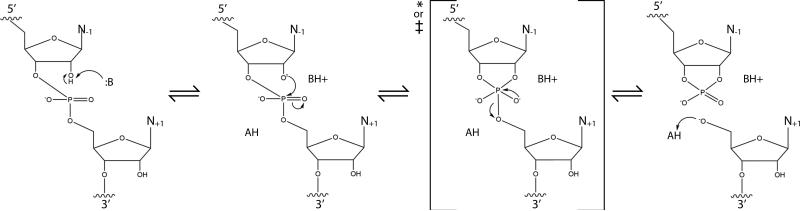
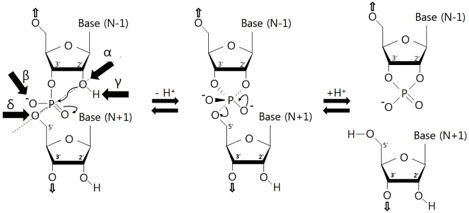
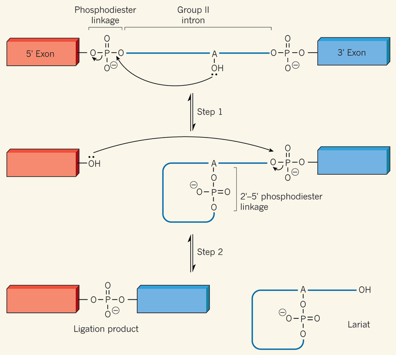
Ce groupe induit de profondes différences entre l'ADN et l'ARN. Principalement parce qu'il rend la molécule d'ARN moins stable chimiquement que la molécule ADN en facilitant les réactions d'auto-clivage :
- La molécule ADN (le génome) est donc mieux adaptée à la conservation de génération en génération de l'information génétique.
- Le caractère transitoire de la molécule d'ARN se prête davantage à un rôle d'adaptateur dynamique aux changements d'état de la cellule.
La chimie prébiotique suppose que l'équilibre entre le clivage et la ligation des ARN a été déterminant :
- dans l'assemblage spontané des premiers ARN en absence de modèle
- dans l'apparition de l'évolution Darwinienne basée sur la compétition entre variants de séquence pour les ressources chimiques
Le groupe 2'-OH est un donneur et un accepteur de liaison hydrogène : il stabilise de nombreux types de paires de bases [non Watson-Crick] (non canoniques) que l'on ne trouve pas dans l'ADN et il facilite ainsi le compactage des hélices d'ARN. L'évolution s'est appuyée sur les propriétés d'auto-interaction de l'ARN pour générer un très grand nombre de structures très diverses capables d'établir des interactions spécifiques ARN - ARN, ARN - protéine, ARN - ADN, ARN - petite molécule ou ARN - ion.
Les différents types d'appariements des paires de bases
Les paires de bases de type Hoogsteen (Karst Hoogsteen) ont des caractéristiques structurales différentes des paires de bases de type Watson - Crick. L'angle entre les 2 liaisons glycosidiques (environ 80° dans la paire A - T) est plus grand et la distance C1' - C1' (environ 8,6 Å) est plus petite. Dans le cas des paires de bases de type Hoogsteen inversé, une base est tournée de 180° par rapport à l'autre.
Une paire de bases de type wobble est un appariement entre 2 nucléotides dans les molécules d'ARN qui ne suit pas les règles des paires de bases de type Watson - Crick. Les 4 principales paires de bases de ce type sont : G - U, hypoxanthine - U, hypoxanthine - A et hypoxanthine - C.
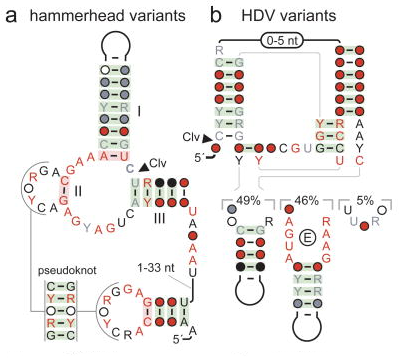
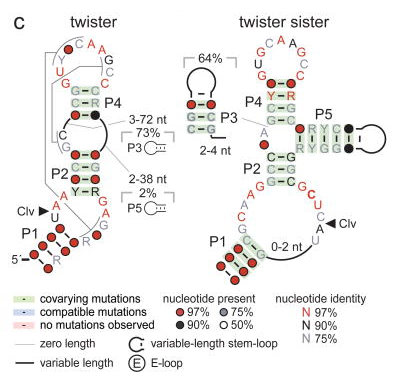
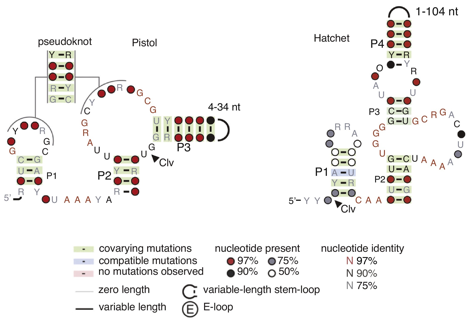
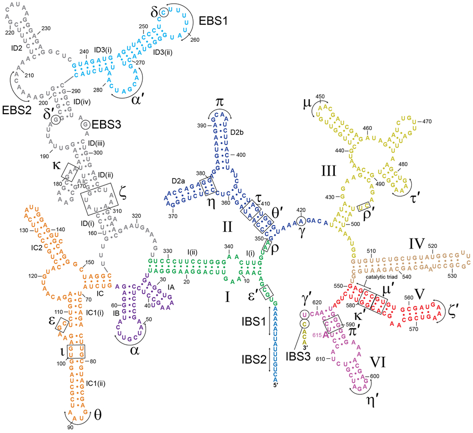
Quelques exemples de structures secondaires et tertiaires de l'ARN
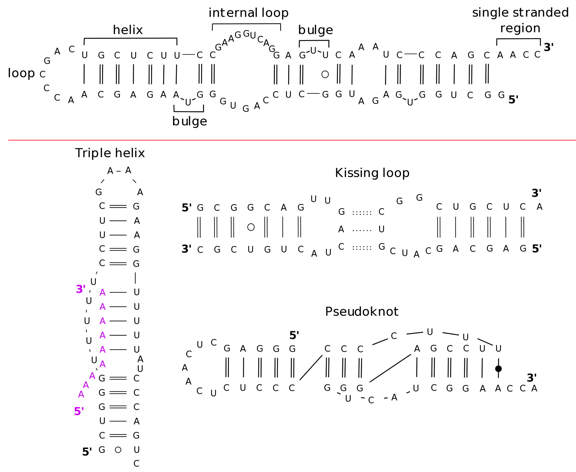
Source : Blythe et al. (2016)
- En haut, petite région située à l'extrémité 5' du long ARN non codant ("long ncRNA" - voir ci-dessous) de B2-SINE.
- "Triple helix" : une région double brin de l'élément d'expression et de rétention nucléaire ("Expression and Nuclear retention Element" - ENE) séquestre la queue poly-A de l'ARN (en magenta) par des interactions de type Watson - Crick et de type Hoogsteen qui aboutissent à une triple hélice stabilisante (PDB 3P22).
- "Kissing loop" : boucle entre le substrat [tige-boucle] et le domaine catalytique [tige-boucle] par le ribozyme Neurospora Varkud satellite (voir ci-dessous - PDB 2MI0). Les 3 paires de bases de type Watson - Crick responsables de l'interaction entre les deux boucles sont en pointillés.
- Pseudonœuds ("pseudoknot") : interactions réciproques entre 2 [tiges-boucles] où la boucle de chaque [tiges-boucles] forme la tige de l'autre (PDB 1A60 - virus de la mosaique jaune du navet). Les paires de bases de type wobble et de type non Watson - Crick sont représentées par des cercles vides et pleins, respectivement.
Les différents niveaux de structure des ARN
La structure de certains types d'ARN peut être décrite par leur structure primaire, secondaire, tertiaire, voire quaternaire comme dans le cas des protéines.
- La structure primaire d'un ARN est la séquence de ses nucléotides.
- La capacité des brins dARN à apparier des bases (canoniques de type Watson - Crick ou non canoniques tel que le wobble G:U) entraîne la formation de structures secondaires,
- Cette formation est principalement médiée par les liaisons hydrogène et l'empilement des bases des nucléotides.
- Ainsi, des nucléotides diversement appariés et non appariés forment des éléments tels que les tiges, les boucles, les renflements, les jonctions et les pseudonœuds.
- Les éléments de structures secondaires s'assemblent à leur tour via des interactions entre boucles particulières ("kissing loop") ou via un empilement coaxial pour former des structures tertiaires des ARN telles que les triplets U°A-U ou les quadruplexes G (structures extrêmement stables formées par des séquences riches en guanine).
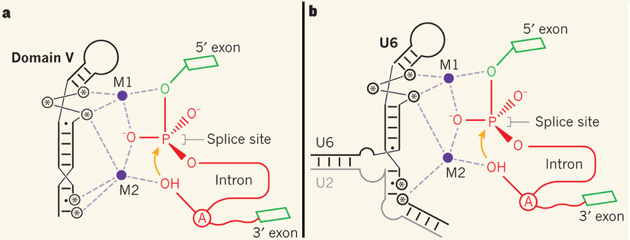
- Enfin, les interactions avec d'autres macromolécules (en particulier ARN-ARN, ARN-ADN et ARN-protéine) conduisent à la formation de structures quaternaires des ARN tels que les ribosomes ou les spliceosomes.
|