Réactions d'oxydo-réduction - Relation de Nernst - NAD(P)+ Chaîne respiratoire - Théorie chimio-osmotique - Synthèse d'ATP |
|
1. Réactions d'oxydo-réduction et potentiel de réduction standard
2. Potentiel de réduction de 2 demi-réactions redox : Relation de Nernst 3. Relation [potentiel de réduction standard - énergie libre de Gibbs standard] 4. Structure du NAD+ et du NADP+ - mécanisme de transfert de l'ion hydrure 5. Rôles biologiques des formes réduites NADH et NADPH 6. La chaîne respiratoire
|
c. Les protéines de la chaîne respiratoire 7. La synthèse d'ATP par phosphorylation oxydative et l'ATP synthase
8. Cours sur la respiration chez les végétaux 9. Quelques inhibiteurs et agents découplants de la respiration 10. Liens Internet et références bibliographiques |
|
|
|
1. Réactions d'oxydo-réduction et potentiel de réduction standard. a. Définitions : réducteur - oxydant - réaction et couple redox. De nombreuses réactions du métabolisme mettent en jeu des transferts d'électrons et bien souvent de protons. Une même molécule va donc être dans un état réduit ou oxydé et ces deux formes de la même molécule s'appelle un couple oxydo-réducteur ou couple rédox. Considérons une molécule A : par convention, les deux formes oxydée et
réduite de A s'écrivent : (Aox / Ared). La réaction d'oxydo-réduction ou réaction redox entre ces deux formes s'écrit : αA Aox + n e- <=> βA Ared
On mesure un potentiel de réduction standard à l'aide d'une pile électrochimique constituée de 2 demi-piles. Chaque demi pile est un couple redox, siège d'une réaction rédox. Quand on mesure le potentiel rédox standard d'un couple rédox, l'une de ces demi-piles sert de référence pour la mesure (de la même manière que la mesure de l'énergie libre de Gibbs nécessite que l'on ait fixé des conditions de référence). Cette demi-pile de référence est l'électrode à hydrogène (qui est le siège de l'oxydation de l'hydrogène) :
Cependant, pour les réactions biologiques, les conditions standard se rapportent à une concentration d'ions H+ de 10-7 M (pH = 7) dans la demi pile de référence. Ainsi,
pour les réactions biologiques, on définit un potentiel
de réduction standard dans les conditions physiologiques : E°'. |
2. Potentiel de réduction de 2 demi-réactions redox : Relation de Walther Hermann Nernst Lorsque les électrodes d'une pile sont réunies par un conducteur extérieur au système, un courant électrique traverse spontanément la pile et parcourt l'ensemble du circuit. a. La réaction d'oxydation qui se produit à l'anode où se trouve le couple oxydo-réducteur (Box / Bred) s'écrit : αB Bred <=> βB Box + n e-
b. La réaction de réduction qui se produit à la cathode où se trouve le couple oxydo-réducteur (Aox / Ared) s'écrit : αA Aox + n e- <=> βA Ared
c. La réaction électrochimique totale dans la pile est la somme des 2 demi-réactions rédox et s'écrit : αA Aox + αB Bred <=> βA Ared + βB Box avec : E°'A > E°'B Le potentiel de réduction de cette réaction globale, ΔE'réaction, est donné par la relation de Nernst :
Voir une démonstration de l'équation de Nernst. Exercices de calculs de potentiels rédox (et autres : thermodynamique, cinétique ...). |
3. Relation entre la différence de potentiel de réduction standard et la variation d'énergie libre de Gibbs standard La différence de potentiel de réduction standard ΔE°' associée au transfert d'électrons d'un couple rédox est liée à la variation d'énergie libre de Gibbs standard par la relation : ΔG°'réaction = - n . F . ΔE°'réaction
|
|
4. Structure du NAD+ et du NADP+ - mécanisme de transfert de l'ion hydrure par les déshydrogénases Dans un trés grand nombre de réactions d'oxydo-réduction qui ont lieu dans la cellule, les électrons sont transférés par des coenzymes : on peut citer à titre d'exemple, le nicotinamide adénine dinucléotide ou NAD+ et le nicotinamide adénine dinucléotide phosphate ou NADP+.
Enfin, les spectres d'absorption des formes oxydée et réduite de ces deux coenzymes sont distincts. Cette propriété est utilisée dans les dosages enzymatiques qui utilisent des déshydrogénases ("dehydrogenase" en anglais) dont un exemple de classification selon l'"Enzyme Commission" (E.C.) est : E.C. 1.-.-.- : classe des oxydoréductases |
5. Rôles biologiques des formes réduites NADH et NADPH Malgré des structures très semblables, les formes réduites de ces deux coenzymes sont employées dans la cellule de manière très différente. Le NADH est produit par des réactions de voies du catabolisme (dégradation) :
Le NADPH est produit par des réactions de voies de l'anabolisme (biosynthèse) telles que la voie des pentoses phosphate.
|
6. La chaîne respiratoire a. Généralités sur la respiration cellulaire aérobie De manière schématique, les réactions du catabolisme ont deux finalités :
La respiration cellulaire correspond donc aux processus suivants :
Source : "Biologie"Campbell (1995) Ed. De Boeck Université Chez les plantes un processus analogue a lieu dans les chloroplastes. Un processus très semblable se déroule chez les bactéries. |
| b. La mitochondrie et la chaîne
respiratoire
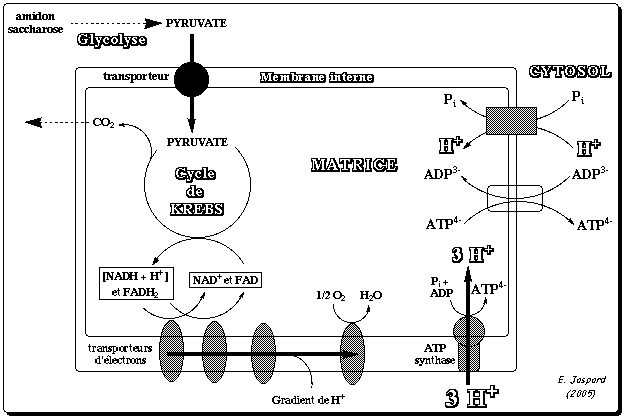
La mitochondrie est limitée par deux membranes caractérisées par des propriétés très différentes : La membrane externe est pauvre en protéines. Elle contient, en particulier, des protéines transmembranaires, les porines, qui permettent le passage des ions et des métabolites hydrosolubles de masse molaire < 7000 Daltons. A l'inverse, la membrane interne est très riche en protéines mais elle est quasiment imperméable aux ions et aux métabolites hydrosolubles. Ces substances ne peuvent traverser la membrane qu'à l'aide de protéines membranaires de transport (l'ATP, l'ADP et le Pi sont transportés par ce type de protéines), ou de systèmes plus complexes qu'on appelle navette. L'espace entre ces deux membranes s'appelle l'espace intermembranaire. La zone interne de la mitochondrie (bordée par la membrane interne) s'appelle la matrice. Elle contient les enzymes du cycle de Krebs et la plupart de celles qui catalysent l'oxydation des acides gras. La chaîne respiratoire est localisée dans la membrane interne des mitochondries. Le nombre des crêtes accroit la surface de cette membrane et ainsi chaque mitochondrie contient des milliers d'exemplaires de la chaîne de transport d'électrons. Les crêtes pénètrent dans la matrice. Certaines protéines mitochondriales sont synthétisées par la mitochondrie (génome mitochondrial), mais la plupart d'entre elles sont codées par le génome nucléaire puis importées dans la mitochondrie. Voir un cours sur la biogénèse (synthèse, adressage et assemblage) des protéines membranaires intégrales. |
c. Les protéines de la chaîne respiratoire La chaîne respiratoire est un ensemble de complexes protéiques qui assurent un transfert de protons et/ou d'électrons comme le ferait "une équipe de rugby qui se passe le ballon". Les caractéristiques des quatre complexes protéiques sont les suivants : |
| Complexe | Protomères | Masse molaire (Da) | Composants |
| complexe I : NADH-coenzyme Q oxydoréductase | 44 (mammifères) |
> 800.000 |
|
| complexe II : succinate-coenzyme Q oxydoréductase | 4 | 125.000 |
|
| complexe III : coenzyme Q-cytochrome c oxydoréductase | 8 | 220.000 |
|
| complexe IV : cytochrome c oxydase | 12 | 200.000 |
|
|
d. Mécanismes du transfert des électrons Aperçu complet et très détaillé de l'ensemble des complexes de la chaîne respiratoire. C'est le complexe protéique constitué du plus grand nombre de sous-unités dont 7 sont codées par le génome mitochondrial. Lien vers la sous-unité 75 kDa. Le (NADH + H+) cède ses électrons par paire (sous forme d'ion hydrure, H-) alors que le coenzyme Q (ou ubiquinone) les accepte un par un. Le complexe I catalyse la réduction de la FMN ou flavine mononucléotide par NADH selon les étapes suivantes : +H+, +
H- -H+ , - e- -H+ , - e- La FMN est donc une sorte de "convertisseur" d'un flux bi-électronique en un flux mono-électronique. Elle cède ses électrons à un ensemble de centres [Fe-S], accepteurs mono-électroniques, qui eux-mêmes les cèdent au coenzyme Q qui est réduit en passant par un intermédiaire semi-quinone : +e- +2H+ , + e-
Le complexe III
|
|
e. Les systèmes de navette ("shuttle") Au cours de la glycolyse, 2 molécules de NAD réduit (NADH) sont formées par molécule de glucose dégradé.
Elle met en jeu 2 enzymes :
Le pouvoir réducteur entre dans la mitochondrie de la manière suivante :
|
|
Elle met en jeu de nombreuses enzymes et transporteurs membranaires : en particulier, la malate déshydrogénase (MDH1 et MDH2 - E.C. 1.1.1.37) et l'aspartate transaminase (GOT1 et GOT2, E.C. 2.6.1.1), présentes à la fois dans le cytosol et dans la mitochondrie. Les principales étapes du mécanisme sophistiqué par lequel le pouvoir réducteur du NADH entre dans la mitochondrie sont les suivantes :
Remarque : chez les organismes anaérobies (ou au cours de la fermentation), il n'y a pas de chaîne respiratoire (ou elle n'est pas employée). La réoxydation du NADH formé au cours de la glycolyse se fait via la conversion du pyruvate en lactate ou en éthanol. |
f. Bilan - résumé de la chaîne de transport d'électrons L'enchaînement des réactions d'oxydo-réduction qui ont lieu lors du transfert des électrons de la chaîne respiratoire est résumé dans la figure ci-dessous. L'énergie libre de Gibbs étant une grandeur d'état thermodynamique, on peut ne considèrer que les réactions impliquant le premier donneur de protons et d'électrons (NADH) et le dernier accepteur d'électrons (l'oxygène moléculaire, O2). Les deux demi-réaction rédox qui, du point de vue énergétique, résument la chaîne respiratoire sont : |
| (1) NADH + H+ -> NAD+ + 2H+ +2 e- | E°'(NAD+/ NADH) = - 0,32 V |
| (2) 1/2 O2 + 2H+ +2 e- -> H2O | E°'(1/2 O2 / H2O) = + 0,82 V |
| Réaction : NADH + H+ + 1/2 O2 -> NAD+ + H2O | E°'(1/2 O2 / H2O) - E°'(NAD+/ NADH) = + 1,14 V |
D'après la relation qui lie la différence de potentiel de réduction standard, ΔE°'réaction, à la variation d'énergie libre de Gibbs standard, ΔG°'réaction : ΔG°'réaction = - n . F . ΔE°'réaction = - (2 . 96500 . 1,14) ≈ - 220 kJ.mol-1 Puisque la variation d'énergie libre standard de la synthèse de l'ATP est environ +30,5 kJ.mol-1, la variation d'énergie libre standard liée à la réoxydation du NADH devrait permettre, en théorie, la synthèse de [220 kJ.mol-1 / 30,5 kJ.mol-1] = "7 molécules d'ATP par molécule de NADH réoxydé", ce qui n'est pas correct. En effet, 3 molécules d'ATP sont synthétisées :
|
7. La synthèse d'ATP par phosphorylation oxydative et l'ATP synthase a. Théorie chimio-osmotique et force proton-motrice La théorie chimio-osmotique formulée par Peter Mitchell en 1961 (Prix Nobel en 1978) postule que le gradient de concentration de protons crée à travers la membrane sert de réservoir d'énergie libre de Gibbs pour la synthèse d'ATP. En d'autres termes, au fur et à mesure que les électrons traversent les quatre complexes de la chaîne de transport d'électrons, des protons passent de la matrice à l'espace intermembranaire et générent un gradient de concentration de protons qui constitue ce réservoir d'énergie libre. En effet, quand ces protons repassent la membrane interne en traversant une protéine membranaire intrinsèque, l'ATP synthase, cette énergie libre est libérée et utilisée par l'ATP synthase pour synthétiser l'ATP. En conséquence, la matrice devient :
que l'espace intermembranaire. La théorie chimiosmotique implique donc que la membrane interne est imperméable aux protons, sinon le gradient se dissiperait au fur et à mesure.
|
c. Le complexe V ou ATP synthase Le complexe V, appelé ATP synthase F0F1 (nom officiel : H+-transporting two-sector ATPase, E.C. 7.1.2.2), utilise le gradient de concentration de protons comme source d'énergie pour synthétiser l'ATP : le sigle "F" désigne un facteur de couplage, selon la nomenclature des enzymes : en effet, l'ATP synthase couple la phosphorylation de l'ADP en ATP à l'oxydation de substrats par la mitochondrie. les ATP synthases sont des protéines oligomériques de masse molaire supérieure à 450 kDa et de structure très complexe : les types différents de chaînes polypeptidiques qui les composent sont au nombre de 8 chez E. coli, 8 ou 9 dans le chloroplaste, 10 ou plus dans la mitochondrie et au moins 13 pour celle du coeur de boeuf.
|
Attention : il ne faut pas confondre l'ATP synthase F0F1 et les ATPases de type "F" (ou F0F1-ATPase). Ces dernières sont des pompes ioniques ou ATPases ionophores : elle hydrolysent l'ATP. L'énergie qu'elles libèrent en hydrolysant l'ATP est utilisée pour le transport d'ions. Il existe plusieurs autres types de pompes ioniques ATPases qui hydrolysent l'ATP.
D'autres types d'ATPases :
|
d. Le mécanisme de la synthèse de l'ATP par l'ATP synthase La réaction peut s'écrire de la manière générale suivante : [H+espace intermembranaire] + [ADP + Pi]matrice -> [H+matrice] + [ATP + H2O]matrice
|
|
α. Structure "rotor-stator / arbre à came" de l'ATP synthase F0F1 L'ATP synthase est ancrée dans la membrane par la sous-unité a de F0 : cette sous-unité constitue le "stator" (la partie fixe) de la "machinerie moléculaire rotative". Les contraintes résultant de cet ancrage sont transmises aux sous-unités b et δ qui relient le complexe hexamérique [α3β3] du moteur F1. Les pièces mobiles sont l'arbre γ central et la sous-unité c10-12 (en forme de disque) qui y est rigidement attachée et qui peut tourner par rapport à la sous-unité a (figure ci-dessous).
source : Kulish et al. (2016) |
source : Wang & Oster (1998) |
|
β. Description du mécanisme des sous-unités de F1 Le composant F1 de l'ATP synthase de E. coli a une stoechiométrie α3β3γδε (chaque lettre indiquant un type différent de sous-unité). F1 est donc schématiquement un cylindre composé de sous-unités α et β altérnées et d'une sous-unité γ située au centre de ce cylindre.
source : The Nobel Prize in Chemistry 1997 |
δ. Le mécanisme structural des sous-unités de l'ATP synthase lors de la synthèse des 3 molécules d'ATP est le suivant :
Source : Université de Leeds Voir des compléments concernant le mécanisme de l'ATP synthase. |
Considérons la molécule d'ATP qui est synthétisée au niveau de la sous-unité dans la conformation βT:
|
9. Quelques inhibiteurs et agents découplants de la respiration |
| 10. Liens Internet et références bibliographiques |
| "Principes de Biochimie" Horton, Moran, Ochs, Rawn et Scrimgeour (1994) - Ed. DeBoeck Universités - ISBN : 2-8041-1578-X | |
|
"ATP synthase web page" Voir une liste de sites consacrés à l'ATP synthase Voir une série d'animations et d'illustrations consacrés à l'ATP synthase |
|
|
Mitchell P. (1961) "Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism" Nature 191, 144 - 148. Boyer et al. (1973) "A new concept for energy coupling in oxidative phosphorylation based on a molecular explanation of the oxygen exchange reactions" PNAS 70, 2837 - 2839 Abrahams et al. (1994) "Structure at 2.8 A resolution of F1-ATPase from bovine heart mitochondria" Nature 370, 621 - 628 Wang & Oster (1998) "Energy transduction in the F1 motor of ATP synthase" Nature 396, 279 - 282 |
|
|
Kulish et al. (2016) "F1 rotary motor of ATP synthase is driven by the torsionally-asymmetric drive shaft" Sci Rep. 6, 28180 Xiao et al. (2018) "NAD(H) and NADP(H) Redox Couples and Cellular Energy Metabolism" Antioxid. Redox Signal. 28, 251 - 272 |
|