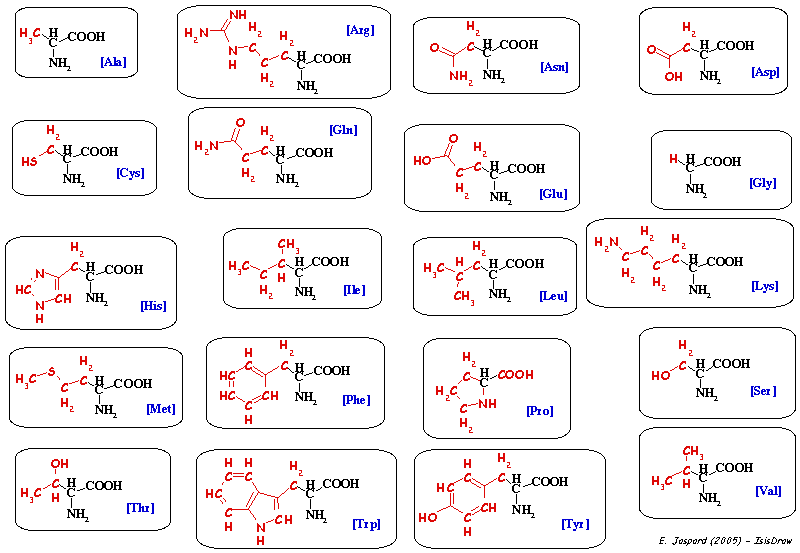
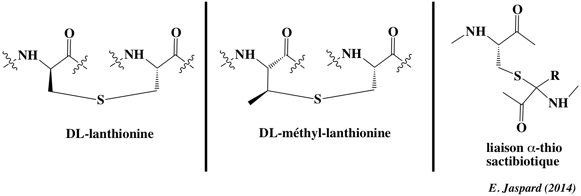
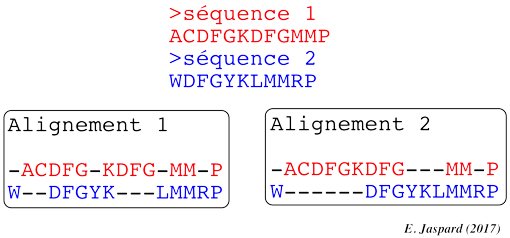
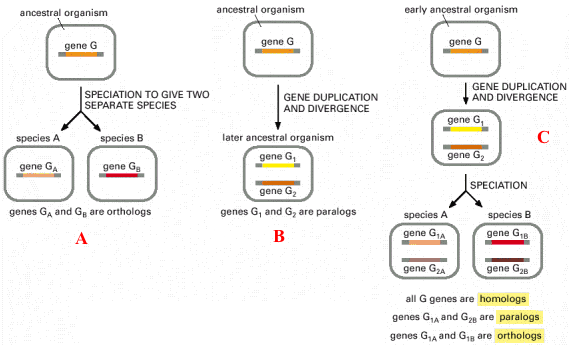
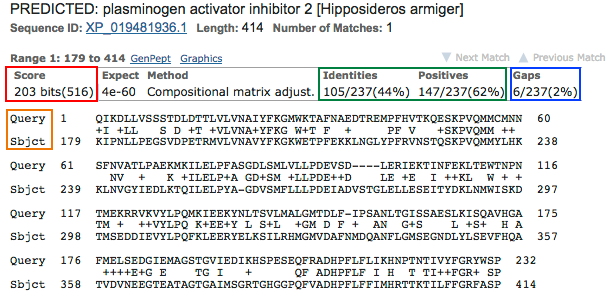
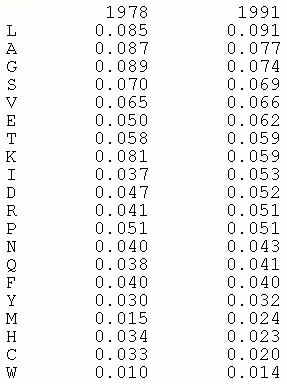
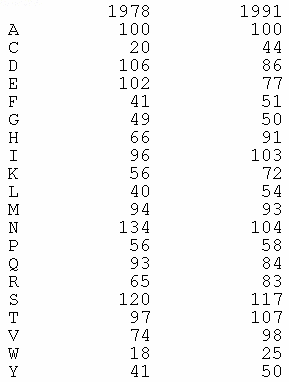
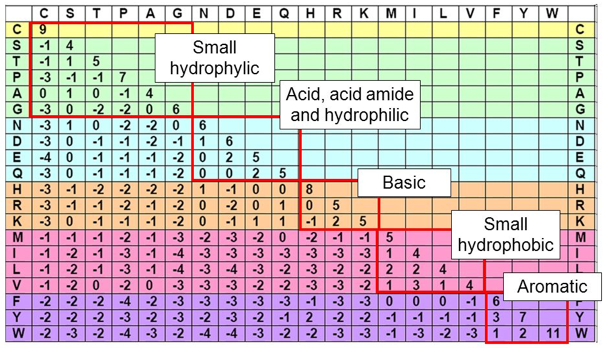
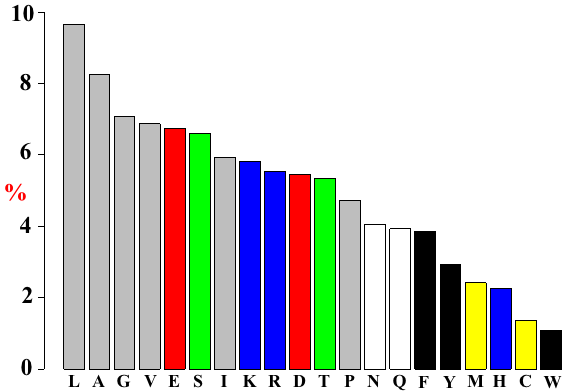
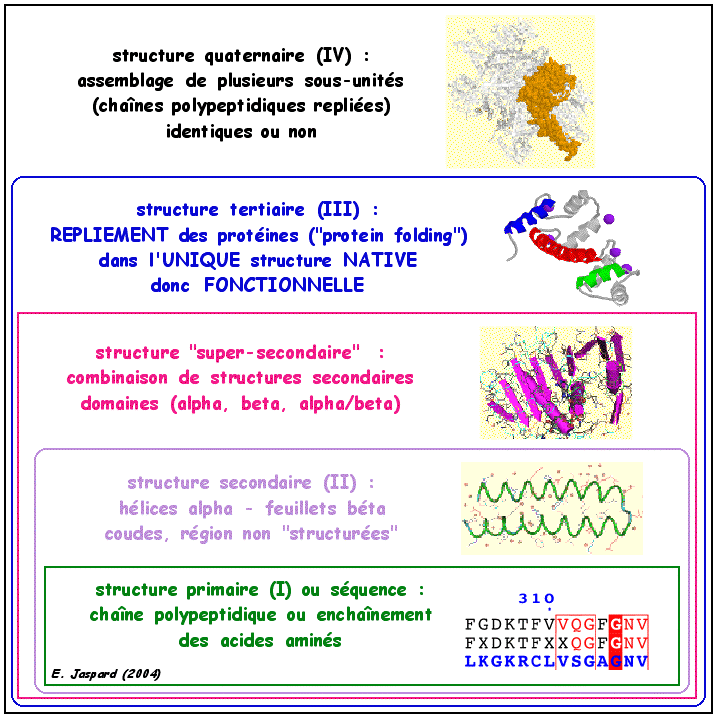
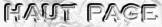1. Structure générale et stéréochimie des acides aminés
A l'exception de la glycine (Gly), tous les acides aminés possèdent un carbone α asymétrique (ou chiral). Il existe donc pour chacun d'entre eux deux isomères stéréochimiques, images en miroir, appelés énantiomères (figure ci-dessous).
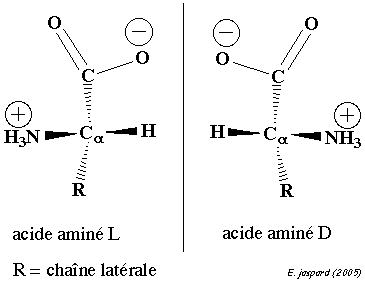
La convention pour définir la stéréochimie du carbone a s'appuie sur celle des énantiomères du glycéraldehyde. A quelques rares exceptions près, les acides aminés constitutifs des protéines sont tous de configuration L.
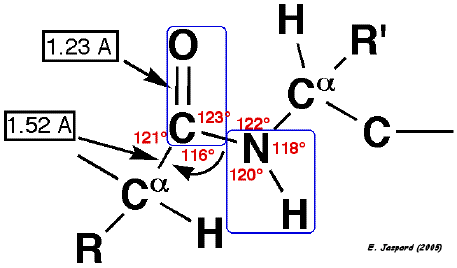
Dans la cellule (pH neutre), le groupe α-carboxyle et le groupe α-aminé sont ionisés : pKa groupe carboxyle : 1,8 à 2,5 - pKa groupe aminé : 8,7 à 10,7
Les acides aminés sont des molécules amphotères (ou zwiterrions).