Bioinformatique : prédiction et analyse des structures des protéines Motifs, profils, signatures, domaines, familles & superfamilles de protéines |
|
1. Introduction et définitions
2. Concepts structuraux issus des séquences en acides aminés
3. Les profils PSSM ("Position Specific Scoring Matrices")
4. Les modèles et les profils de Markov cachés (HMM)
5. Le consortium de bases de données InterPro
|
6. Analyse de quelques propriétés physico-chimiques des acides aminés
7. Structures des protéines 8. La base de données des structures de macromolécules biologiques PDB
9. Modélisation de structures de protéines par homologie
10. Le programme MODELLER
11. Mécanique moléculaire appliquée à la structure des protéines 12. Liens Internet et références bibliographiques |
1. Introduction et définitions a. Information issue des domaines en omique Les domaines en omique engendrent une quantité inouïe d'information, en particulier ceux qui génèrent des données de séquences de nucléotides (génomique, transcriptomique) ou de séquences d'acides aminés (protéomique). On ne peut pas analyser cette information en temps réel. Par ailleurs, décrypter la fonction d'une protéine ou d'une enzyme nécessite :
Une analyse aussi détaillée et complète nécessite des mois, voire des années pour chaque protéine. C'est impossible d'effectuer ce travail pour les millions de protéines (tous organismes confondus) dont on suppose avoir déterminé l'existence par le biais du séquençage (voir le tableau ci-dessous). |
| Type de données biologiques | Base de données | Nombre |
Séquences nucléotidiques Nombre total de nucléotides |
GenBank (février 2025) | ≈ 256 millions ≈ 5416 milliards (ou 5,42 billions) |
|
Séquences protéiques annotées par programmes Nombre total d'acides aminés |
UniProtKB/TrEMBL (février 2025) TrEMBL ("Translated EMBL Nucleotide Sequence Database") |
≈ 253 millions ≈ 90 milliards |
|
||
| Séquences protéiques annotées manuellement ("reviewed") | UniProtKB/Swiss-Prot (avril 2025) | ≈ 573.000 |
| Séquences de protéines de référence | RefSeq (mars 2025) | ≈ 400 millions |
| Structures 3D de protéines : Déterminées expérimentalement Prédites (AlphaFold) |
PDB ("Protein Data Bank") (avril 2025) | ≈ 234.000 ≈ 1,07 millions |
|
||
| Domaines non-redondants | SCOP2 ("Structural Classification of Proteins") (avril 2024) | ≈ 72.600 |
| Familles de protéines | > 24.000 Pfam-N |
|
|
||
| Nombre de repliements ("protein folds") (n'inclue pas les repliements de protéines artificielles générées par apprentissage profond) |
SCOP2 (avril 2024) | 1562 |
|
||
b. Analyse bioinformatique de la relation structure - fonction des protéines L'enchaînement séquence => structure => fonction traduit qu'une séquence en acides aminés contient l'information nécessaire pour que la chaîne polypeptidique se replie dans la conformation native qui confère à la protéine (à l'enzyme) sa fonction biologique (son activité enzymatique).
Cet enchaînement, démontré par les travaux de Christian Anfinsen (Prix Nobel 1972), est vrai pour une très grande majorité des protéines (enzymes). Cependant, d'autres processus qui contribuent au repliement de certaines chaînes polypeptidiques ont été mis en évidence depuis plusieurs années.
|
Prédiction des propriétés structurales et fonctionnelles des macromolécules biologiques Compte-tenu du nombre colossal de macromolécules biologiques à étudier, de la multiplicité et de la diversité des propriétés à décrire, des moyens méthodologiques requis et leur coût, il est impossible d'analyser expérimentalement cet ensemble de données dans un laps de temps raisonnable. En conséquence, les méthodes bioinformatiques de prédiction des propriétés structurales et fonctionnelles des macromolécules biologiques sont indispensables. Le mot prédiction est capital :
Enfin, l'évolution des technologies informatiques est parallèle au développement des méthodes de prédiction :
|
Apport magistral de l'apprentissage automatique L'apprentissage profond (ou "deep learning") s'appuie sur :
L'apprentissage profond révolutionne la notion de prédiction : les informations que l'on calcule sont certes encore prédites mais leur "mimétisme avec la réalité" est de plus en plus important, diminuant ainsi la part de hasard et/ou d'incertitude. Voir un développement de l'apprentissage profond appliqué au "langage protéique". L'exemple phare dans le domaine de la prédiction des structures de protéines est le développement du programme AlphaFold (DeepMind - Google) qui :
Voir un développement de l'intelligence artificielle appliquée à la structure 3D des protéines. |
2. Concepts structuraux issus des séquences en acides aminés a. Définitions de différents concepts dans la base de données Uniprot On décompose les structures des protéines en entités structurales de différentes tailles en acides aminés, en entités associées à une fonction, à une particularité évolutive ou à une propriété conformationnelle, ...
|
| Entité structurale | Caractéristiques & exemples |
| Motif ("motif") | Un motif est une courte séquence d'acides aminés associée à une fonction commune (exemples : la liaison aux protéines ou l'adressage à un compartiment subcellulaire) d'une famille de protéines.
Exemples:
|
| Répétition ("repeat") | Les répétitions varient :
Une explication du succès évolutif des répétitions est leur faible "coût" énergétique cellulaire : les protéines de grande taille et thermodynamiquement stables peuvent provenir de duplications intragéniques plutôt que de processus complexes de création de novo d'hélice(s) α et de feuillet(s) β. |
| Biais de composition ("compositional bias") | Deux types de régions à biais de composition :
Exemple : "Another Transcription Unit protein" (ATU) => biais Arg/Ser-rich (RS domain) et Gly/Ser-rich |
| Bobines enroulées ("Coiled coil") | Elles sont constituées de 2 (ou plus) hélices α enroulées l'une autour de l'autre pour former un super-enroulement ("supercoil").
Dans UniProtKB, l'annotation des bobines enroulées est prédite avec le programme COILS :
|
| Région ("region") | Décrit les caractéristiques d'une région définies expérimentalement :
|
| Domaine ("domain") | Les domaines protéiques sont un concept structural clé pour décrire la structure et la fonction des protéines et regrouper les protéines en familles.
Ils sont présents individuellement ou au sein d'architectures complexes de protéines multidomaines qui évoluent par accrétion de domaines, perte de domaine(s) ou recombinaison de domaines. Les domaines protéiques peuvent donc être considérés comme des "pièces de Lego®". Exemples de noms de domaines protéiques : ANK repeat, annexin, bait region, bromodomain, CBS domain, coiled coil, collagen, EGF-like domain, homeobox, immunoglobulin domain, Kelch repeat, Sh2-domain, transit peptide, zinc-finger, ...
|
| Domaines topologiques ("topological domain") |
|
| Source : Uniprot - "Family and Domains section" | |
b. Séquences des protéines : alignements, motifs, profils et signatures Les alignements de séquences multiples permettent d'identifier les résidus d'acides aminés conservés dans des protéines homologues apparentées de manière lointaine (exemple : protéines ribosomales d'Eucaryotes et de Procaryotes).
|
Les signatures ("fingerprint")
Les signatures sont des modèles mathématiques construits à l'aide de différentes algorithmes et outils informatiques :
Les signatures protéiques sont utilisées pour :
L'emploi des signatures pour identifier la fonction d'une protéine est souvent plus sensible que les recherches de similarité entre paires de séquences comme BLAST. |
c. Les domaines structuraux des protéines Un domaine d'une protéine est une une partie de la chaîne polypeptidique qui se replie indépendemment du reste de la chaîne polypeptidique et qui est associée à une (des) fonction(s) de cette protéine. On peut considérer le domaine protéique comme l'unité de base de la structure des protéines. Voir un cours sur le repliement des chaines polypeptidiques. Les domaines des protéines ont des tailles en acides aminés qui ont des limites :
Longueurs moyennes des domaines des protéines des 3 règnes :
Source : Yadav et al. (2022) Nombre de domaines protéiques dans la base de données Pfam (InterPro) :
|
Lien entre domaines protéiques et repliement des protéines Le repliement d'une chaîne polypeptidique (structure tertiaire) résulte du repliement de son/ses domaine(s).
Source : Schaeffer & Daggett (2011) |
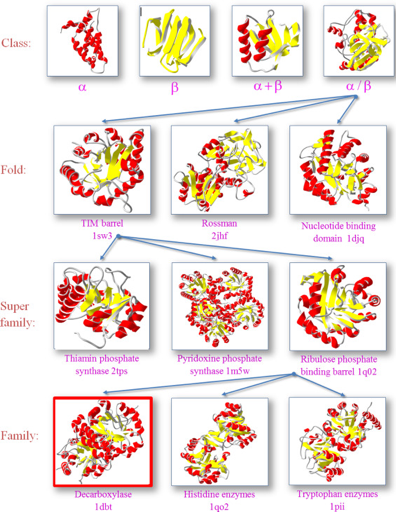
Les différents repliements ("fold") des protéines peuvent être répartis classés en 4 classes principales en fonction du contenu en structures secondaires prédominantes au sein de leur(s) domaine(s).
Voir la base de données de classes de structures des protéines SCOP. Source : Wiltgen M. (2019) |
 |
| Exemples de domaines | Caractéristiques & exemples |
| domaine en faisceau d'hélices ("helix bundle domain") | Un des domaines protéiques les plus fréquents (exemple 1MFT). Dans certains cas, les hélices internes au faisceau, reliées par des boucles, sont regroupées et forment un noyau hydrophobe. |
| domaine en tonneau TIM ("TIM barrel domain") | La Triose phosphate IsoMérase (TIM) est une enzyme dimèrique de la glycolyse. La TIM ne possède qu'un domaine qui est le modèle du repliement dit "en tonneau TIM". C'est un repliement des protéines très répandu constitué de brins de feuillets β parallèles, reliés par des boucles et des hélices. |
| domaine de fixation des nucléotides ("Rossmann fold") |
|
| Protéines à plusieurs domaines | |
| La pyruvate kinase | Cette enzyme de la glycolyse (code 1PKN) est constituée de 3 domaines qui adoptent des repliements différents : 1 domaine en feuillets β et 2 domaines de type α/β. |
| La titine (ou connectine) | C'est l'une des plus grosses protéines connues (masse molaire ≈ 3,82 millions Da chez l'homme) : elle est composée de 132 copies du domaine fibronectine de type III et de 112 copies du domaine immunoglobuline de type II. |
3. Les profils PSSM ("Position Specific Scoring Matrices") En anglais, il existe plusieurs dénominations de ce type de matrices de scores : matrice de pondération de position ("Position Weight Matrix" - PWM), matrice de pondération spécifique de la position ("Position-Specific Weight Matrix" - PSWM) ou matrice de notation spécifique de la position. Dans un profil PSSM, les scores de substitution des acides aminés sont calculés pour chaque position au sein d'un alignement de séquences multiples :
Les scores des profils PSSM sont :
Exemples d'applications des matrices PSSM : localisation sub-cellulaire des protéines, modifications post-transcriptionnelles et post-traductionnelles, prédiction de la structure 2D et 3D des ARN et des protéines, identification des régions de liaison protéine-ADN et protéine-ARN, ... |
b. Principe de la construction d'un profil PSSM
Etapes de la construction d'un profil PSSM
Première étape
Deuxième étape
où i ∈ (1, ..., n) et j ∈ (1, ..., l) ; k est l'ensemble des 20 caractères de l'alphabet des acides aminés (code à 1 lettre) ; I(a=k) est une fonction indicatrice. Troisième étape
|
Illustration
Le profil PSSM le plus probable est DKDYKD. |
c. Le programme PSI-BLAST et le profil PSSM Les matrices ou profils PSSM peuvent être générés, notamment, par le programme PSI-BLAST ("Position-Specific Iterative Basic Local Alignment Search Tool") qui :
Source : Ozsari O. (GitHub) 1. La séquence "requête" ("Query Sequence") est utilisée par le programme PSI-BLAST :
2. Le programme PSI-BLAST génère un premier profil PSSM à partir de cet alignement de séquences multiples. 3. Ce premier profil PSSM est utilisé pour rechercher de nouvelles séquences dans la base de données interrogée avec la même séquence requête : les séquences détectées (au-dessus d'un seuil de score spécifié) lors de cette 2ème itération sont à leur tour ajoutées à l'alignement de séquences multiples et le profil PSSM est affiné.
PSI-BLAST permet ainsi de détecter des relations entre protéines distantes sur le plan évolutif. |
d. Le programme RPS-BLAST et le service Web "CD-Search" Alternativement, les matrices PSSM peuvent être récupérées à partir de la base de données CDD ("Conserved Domain Database" au NCBI) puisque chaque domaine conservé est représenté par un profil PSSM qui traduit les substitutions observées dans les alignements des séquences multiples "souches" ("seed alignments"). Ces profils PSSM de domaines conservés peuvent être recherchés :
Le service Web "CD-Search" (NCBI) fournit une interface Web qui inclue :
|
|
4. Les modèles et les profils de Markov cachés (HMM) Les modèles de Markov cachés ("Hidden Markov models" ou HMM) sont des modèles statistiques puissants utilisés pour convertir plusieurs alignements de séquences en systèmes de notation spécifiques de la position :
a. Principe de construction des profils HMM Lors de la recherche d'homologues distants, il faut disposer du maximum d'informations sur la séquence de la protéine requête et celles de la base de données fouillée pour distinguer les vrais positifs des faux positifs et ainsi obtenir des alignements optimaux. La comparaison [profil - séquence] ou la comparaison [profil - profil] est parfois plus performante (en améliorant la sensibilité et la sélectivité) que la comparaison [séquence - séquence]. Les profils HMM :
Illustration : alignement [HMM séquence requête - HMM séquence cible] avec la suite logicielle HH-suite
Source : Steinegger et al. (2019)
Les états M et I de l'un des HMM ne peuvent être alignés qu'avec les états M ou I de l'autre HMM. Par ailleurs, un état D ne peut être aligné qu'avec un état D ou avec un "gap". Le score de co-émission est la somme des scores de similitude des colonnes des profils alignés, c'est-à-dire les états de la paire MM moins les pénalités spécifiques à la position pour les insertions - délétions. L'alignement est représenté par le chemin rouge le long des deux HMM et la séquence d'état des paires correspondantes est [MM - MM - MI - MM - MM - DG - MM]. Ressources
|
b. Illustration : identification d'un site d'épissage 5' exon - intron Soit une séquence d'ADN qui commence par un exon, contient un site d'épissage 5'SS ("5' splicing site") et finit par un intron (figure ci-dessous). La position du site d'épissage n'est pas connue.
On peut construire un profil HMM à partir de ces informations :
Source : Eddy S.R. (2004) |
|
Le programme HMMER est utilisé pour la recherche des séquences homologues dans certaines bases de données (exemples : Uniprot, PDB, Ensembl, MEROPS, ChEMBL, ...) et la construction d'alignements.
HMMER renvoie 2 jeux de "coordonnées" de domaine pour chaque occurence ("match") avec un profil HMM :
Les alignements complets de Pfam contiennent les coordonnées d'enveloppe de HMMER :
HMMER ("Biosequence analysis using profile hidden Markov Models") : interface Web du programme HMMER à l'EBI. |
5. Le consortium de bases de données InterPro InterPro permet l'analyse de séquences de protéines en les classant dans des familles et en prédisant la présence de domaines et de sites fonctionnels.
Source : InterPro
|
a. Contenu et caractéristiques de Pfam (consortium InterPro) La caractérisation du ou des domaines d'une protéine permet d'en décrypter la/les fonction(s). En effet, la possibilité de diverses combinaisons de multiples domaines explique :
La base de données Pfam est une collection de plus de 24.000 familles de protéines :
|
Types d'entrées de la base de données Pfam Les entrées de Pfam sont classées en 6 catégories, en fonction de la longueur et de la nature des parties de la séquence incluses dans l'entrée :
Source : Course Online - EBI Un profil HMM unique est souvent insuffisant pour modéliser une superfamille de protéines : les entrées liées à l'évolution sont combinées pour former de plus vastes ensembles appelés clans. L'inter-relation de ces familles est définie par :
|
Méthode de construction des alignements Pfam Chaque famille Pfam est constituée d'un alignement de séquences multiples généré de la manière suivante :
Quelques programmes pour gérer les fichiers Pfam :
|
b. Illustration : le profil HMM du domaine EF-hand_1 α. Via l'interface web de Pfam A partir du lien "Signature" à gauche de la page EF-hand_1 (PF00036), on obtient la figure - appelée "logo"- ci-dessous :
Source : Pfam
A partir de cette page, cliquer sur le lien "Curation & model". En cliquant sur le lien "Download the raw HMM for this family" en bas, on obtient le fichier du profil EF-hand_1 (longueur 29 acides aminés). β. Récupération d'un fichier HMM de Pfam avec un script Python
Interface web pour récupérer un fichier de profil HMM de Pfam. |
6. Analyse de quelques propriétés physico-chimiques des acides aminés Les acides aminés ont des propriétés physico-chimiques trés diverses. La base de données " ProtScale" fournit près de 60 tables de valeurs de ces propriétés. En voici quelques exemples (base de données "ProtScale") :
Voir une liste de tables de 566 propriétés physico-chimiques. |
a. Hydrophobicité d'une séquence avec une fenêtre glissante La valeur d'hydrophobicité de chaque résidu d'acide aminé dans la séquence est calculée en tenant compte de l'indice d'hydrophobicité du résidu considéré mais également de celui d'un certain nombre d'acides aminés en amont et en aval qu'on désigne par "fenêtre". Cette approche permet de tenir compte de l'incidence des acides aminés contigüs à chaque acide aminé. En revanche il ne tient pas compte des acides aminés éloignés dans la séquence mais proches dans l'espace après le repliement de la chaine polypeptidique.
Exemple de calcul avec une séquence IVLFCMAG et une fenêtre glissante de 3 acides aminés :
Exemple : récepteur muscarinique de l'acétylcholine M1 (P11229) - échelle de Kyte & Doolittle
Figure A : fenêtre n = 7 - figure B : fenêtre n = 21
Applications
|
b. Amphiphilicité de structures secondaires et moment hydrophobe Cette propriété physico-chimique est liée à la répartition des résidus hydrophiles et hydrophobes sur une face d'une structure secondaire. La figure ci-dessous illustre la prédiction de formation d'une hélice α de classe A pour les acides aminés 144-179 de la protéine LEAP ("Late Embryogenesis Abundant Protein" - Q5NJL5).
Prédiction effectuée avec le programme HeliQuest en utilisant une fenêtre de 36 acides aminés. En bleu et rouge : résidus d'acides aminés chargés (K, R, D, E). En jaune et gris : résidus d'acides aminés non polaires. La flèche indique le moment hydrophobe. Pour quantifier l'amphiphilicité de structures secondaires des protéines, Eisenberg et al. (1982) ont développé la notion de moment hydrophobe, μ(θ), qui mesure la répartition de résidus d'acides aminés hydrophiles et hydrophobes dans une structure régulière de période θ. Ainsi, pour une structure secondaire constituée de n résidus d'acides aminés consécutifs, la relation générale de μ(θ) est :
La largeur optimale de la fenêtre est n = 11 résidus d'acides aminés pour une hélice α transmembranaire avec θ = 102°.
|
c. Surface accessible au solvant et surface exposée au solvant La surface des acides aminés des protéines accessible au solvant est un paramètre déterminant pour l'étude du repliement des chaînes polypeptidiques et le calcul de leur stabilité. On fait "rouler" une molécule d'eau (considérée comme une sphère) autour des atomes de la protéine, considérés également comme des sphères de rayon égal à leur rayon de van der Waals. Le centre de la molécule d'eau est à une distance du centre de chaque atome qui est égale à la somme du rayon de van der Waals de la molécule d'eau (1,4 Å) et du rayon de van der Waals de l'atome.
Voir la partie du guide d'utilisation du logiciel Chimera dédié au calcul la surface moléculaire. |
d. Rappels sur les différents types d'hélices Les hélices α Deux hélices α adjacentes sont généralement arrangées de manière antiparallèle. Elles sont compactées par les liaisons hydrogène qui s'établissent entre les chaînes latérales des acides aminés. Ces paires (ou unités) sont souvent arrangées en faisceau à 4 hélices dans lequel les chaînes latérales des 4 hélices α sont empilées et forment un coeur hydrophobe au centre du faisceau. Les faisceaux à 4 hélices forment des domaines α dans les protéines. La myohémérythrine, le cytochrome b562, la petite protéine Rop qui se lie à l'ARN, sont des protéines de ce genre. Les autres types d'hélices Hélice 310
|
| type de structure | valeurs des angles (degrés) | nombre moyen de résidus par tour | translation par résidu (Å) | ||
| Φ | Ψ | ω | |||
| hélice α | - 57 | - 47 | 180 | 3,6 | 1,5 |
| hélice 310 | - 49 | - 26 | 180 | 3,0 | 2,0 |
| hélice π | - 57 | - 70 | 180 | 4,4 | 1,15 |
| hélice polyproline I | - 83 | + 158 | 0 | 3,38 | 1,9 |
| hélice polyproline II | - 78 | + 149 | 180 | 3,0 | 3,12 |
| Source : base de données PROWL -> consulter l'item : "Residue hydrogen bonding" Voir la méthode du diagramme de Ramachandran pour une définition des angles. |
|||||
|
a. Techniques pour déterminer la structures des protéines Voir un cours sur les différentes méthodes (théoriques et pratiques) pour déterminer la structure des macromolécules biologiques en général et des protéines en particulier. b. Bases de données liées aux structures des protéines Il existe un grand nombre de bases de données liées aux structures des protéines. Ces ressources classent les protéines en fonction de leur degré de similarité, mais diffèrent par les définitions qu'elles emploient et leurs méthodes de classification. Il y a cependant un consensus général en ce qui concerne l'ordre hiérarchique entre topologie globale ou repliement, superfamille, famille et domaines individuels. De nombreuses protéines ayant la même topologie auront évolué de manière convergente, mais les membres des superfamilles et des familles sont susceptibles d'être issus d'un ancêtre commun par une évolution divergente.
|
8. La base de données de structures des macromolécules biologiques PDB ("Protein Data Bank") Généralités La base de données PDB RCSB ("Research Collaboratory for Structural Bioinformatics") est membre du consortium "worldwide PDB" (wwPDB - constitué de PDB (USA), PDBe(Europe) et PDBj (Japon)). Un code d'accession PDB est constitué de 4 caractères (1 chiffre puis 3 caractères qui peuvent être un chiffre ou une lettre en majuscule). Il y a 419.904 codes possibles d'identification PDB (valeur qui peut augmenter à 466.560 avec le chiffre 0 comme premier caractère). Les quelques 160.000 fichiers (février 2020) représentent environ 38 % des codes d'identification disponibles. Exemple de quelques codes d'accession PDB "parlants" : 1MBN : myoglobine (1973) Signification de quelques champs d'un fichier PDB SSBOND, LINK, ... : annotations de connectivité CRYST1 : paramètres de la cellule unitaire, groupe d'espace et valeur de Z ORIGXn, SCALEn : transformation à partir des coordonnées orthogonales MODEL : numéro du modèle dans un fichier qui contient plusieurs structures ENDMDL : fin de chaque modèle MODRES : modifications de résidus standards HET : résidus non standard (et ligands, ions et molécules d'eau) HETNAM : nom chimique complet du résidu HETSYM : synonymes du nom du résidu FORMUL : formule chimique du résidu Ressources additionnelles qui décrivent des catégories de molécules particulières :
|
a. Unité asymétrique d'une maille cristalline et cellule unitaire L'unité asymétrique est le plus petit volume d'une structure cristalline (maille cristalline) auquel les opérations de symétrie du groupe d'espace du cristal considéré peuvent être appliquées afin de reconstituer la cellule unitaire ("unit cell" - l'unité de répétition du cristal) complète. Quand la cellule unitaire est répliquée dans les 3 dimensions, on reconstitue le cristal entier. Exemple ci-dessous :
Source : PDB Remarque : le volume de l'unité asymétrique est donc inférieur à celui de la maille du cristal sauf pour le groupe d'espace triclinique P1 dont l'unité asymétrique a un volume égal à celui de la maille. Les opérations de symétrie les plus couramment appliquées aux cristaux de macromolécules biologiques sont les rotations, les translations et les "tour de de vis" ("screw axes", combinaisons [rotation - translation]). |
|
L'assemblage biologique (ou unité biologique - "biological assembly") est l'assemblage macromoléculaire qui est ou semble être la forme fonctionnelle de la molécule. Par exemple, la forme fonctionnelle de l'hémoglobine est constituée de 2 x 2 chaînes polypeptidiques (α2β2). Selon la structure cristalline, des opérations de symétrie peuvent être nécessaires pour obtenir l'assemblage biologique complet. A l'inverse, un sous-ensemble des coordonnées cristallographiques peut suffire pour représenter l'assemblage biologique. En conclusion, un assemblage biologique peut être construit à partir :
Exemples
Outils d'analyse des assemblages biologiques Des bases de données spécifiques, telles que PISA ("Protein Interfaces, Surfaces and Assemblies"), permettent d'analyser les assemblages biologiques de la PDB. jsPISA est un outil web interactif pour le calcul des surfaces macromoléculaires et des interfaces, pour l'évaluation de leurs propriétés et pour l'inférence d'assemblages macromoléculaires probables à partir de données de coordonnées (généralement cristallographiques). |
c. Les fichiers au format mmCIF ("macromolecular Crystallographic Information Format") Un fichier au format mmCIF contient les instructions pour générer un assemblage biologique. Un grand nombre de programme de visualisation moléculaire (dont Jmol) sont compatibles avec ce format. Voir un exemple : fichier 2NBT.cif - toxine de serpent. Un fichier mmCIF contient donc les informations concernant les éléments structuraux qui générent un assemblage biologique. Ces informations se trouvent dans des catégories (ou listes d'informations) appelées "pdbx_struct_assembly", "pdbx_struct_assembly_gen" et "pdbx_struct_oper_list" :
La catégorie "struct_biol" contient les remarques spécifiques des auteurs relatives aux assemblages biologiques. Un dictionnaire de données archive les expériences de cristallographie de petites molécules et leurs résultats. Voir le fichier "mmcif_pdbx.dic" ("PDB Exchange Dictionary - PDBx/mmCIF"). Le format de ce dictionnaire et les fichiers de données basés sur ce dictionnaire sont conformes à la représentation des données appelée STAR ("Self Defining Text Archive and Retrieval"). Extrait d'un fichier au format mmCIF _pdbx_struct_assembly.id 1 _pdbx_struct_assembly.details author_and_software_defined_assembly _pdbx_struct_assembly.method_details PISA _pdbx_struct_assembly_gen.assembly_id 1 _pdbx_struct_assembly_gen.asym_id_list A,B,C,D,E,F,G,H loop_ _pdbx_struct_assembly_prop.biol_id 1 'ABSA (A^2)' 3840 ? loop_ _pdbx_struct_oper_list.id _pdbx_struct_oper_list.matrix[1][1] 1 'identity operation' 1_555 1.0000000000 0.0000000000 2 'crystal symmetry operation' 4_565 1.0000000000 0.0000000000 1_555 : décrit l'opérateur de symétrie utilisé (désigné par le nombre 1) et les opérations de translation nécessaires (le chiffre 555). Les opérateurs de symétrie sont définis par le groupe d'espace et les opérations de translation sont indiquées pour les 3 axes de la cellule unitaire (a, b et c) :
4_565 : utilisation de l'opérateur de symétrie 4, suivie d'une opération de translation de la cellule unitaire dans la direction positive selon l'axe b. |
9. Modélisation de structures de protéines par homologie ("Homology modeling") Les algorithmes de comparaison de structures 3D de deux protéines peuvent être classés en plusieurs catégories :
|
a. Les scores de distances inter-atomiques entre des structures comparées La modélisation par homologie de structures nécessite de superposer les structures des protéines qui sont comparées pour établir la correspondance spatiale entre les acides aminés équivalents dans ces structures. Le score RMSD et le score RMSD local Pour mesurer ces distances inter-atomiques on utilise fréquemment une grandeur appelée écart quadratique moyen (RMSD - "Root Mean Square Deviation", en Å) entre les coordonnées spatiales des atomes des acides aminés appariés. Par exemple, si on considère n atomes de 2 structures, on compare les coordonnées (xi, yi, zi) d'un atome i de l'une de ces structures aux coordonnées (x'i, y'i, z'i) d'un atome i apparié de l'autre structure :
On superpose ainsi les structures en minimisant la valeur de RMSD.
Limites du score RMSD
Pour effectuer un choix pertinent des acides aminés à comparer, on calcule un RMSD local avec les carbones α des acides aminés inclus dans une fenêtre de longueur L+1 :
Le score GDT Le score GDT ("Global Distance Test") est aussi une mesure de la similarité entre 2 structures protéiques qui ont des structures 3D différentes. Ce score est calculé sur la base du plus grand ensemble de carbones α des résidus d'acides aminés de la structure experimentale dont la position dans l'espace correspond (selon un seuil donné : 1 Å, 2 Å, 4 Å et 8 Å) à celles des résidus d'acides aminés de la structure prise pour modèle. Les scores GDT sont les principaux critères d'évaluation des résultats de prédiction issus de l'expérience CASP ("Critical Assessment of Structure Prediction") :
Le score TM Le score TM ("Template Modeling score") est également une mesure de la similarité entre 2 structures protéiques qui ont des structures 3D différentes. Le score TM est une mesure plus précise de la qualité des structures protéiques que le score RMSD et le score GDT souvent utilisés. Dans le calcul du score TM, les distances faibles ont un poids plus élevé que les distances fortes : ce score est donc insensible aux erreurs de modélisation locale.
Exemples de comparaisons et de scores de comparaisons Figure ci-dessous : superpositions des structures de 2 protéines avec différents algorithmes (CE - "Combinatorial Extension of the optimal path", SAL, DALI et TM-align).
L est le nombre de résidus d'acides aminés alignés. Les scores RMSD et TM sont indiqués.
Source : Zhang & Skolnick (2005) Le score lDDT Le score lDDT ("local Distance Difference Test") mesure la façon dont l'environnement dans une structure référence est reproduit dans une structure modèle. Ce score est calculé sur toutes les paires d'atomes dans la structure de référence à une distance inférieure à un seuil prédéfini (appelé rayon d'inclusion) et n'appartenant pas au même résidu d'acide aminé. Ces paires d'atomes définissent un ensemble de distances locales L :
Pour un seuil donné, la fraction des distances conservées est calculée. Le score IDDT est la moyenne de 4 fractions calculées en utilisant des seuils identiques à ceux utilisés pour calculer le score GDT (voir ci-dessus). Divers
|
|
b. Les modules Bio.PDB de BioPython Les modules Bio.PDB de BioPython permettent d'effectuer des calculs de distances et d'autres calculs (surface accessible au solvant, ...) liés aux structures des macromolécules de manière générale. Le module Bio.PDB.PDBParser() permet d'effectuer les opérations suivantes :
import Bio.PDB
from Bio.PDB.PDBParser import PDBParser
decoupage = PDBParser() # creation d'un objet PDBParser
structure = decoupage.get_structure("nom", "fichier.pdb")
modele = structure[0]
Créer un objet structure à partir d'un fichier PDB :
Créer un objet structure à partir d'un fichier mmCIF :
|
| 1er exemple de calcul : la clathrine | 2ème exemple de calcul : la toxine de serpent |
Code PDB : 1XI4 Chaînes polypeptidiques du manteau de clathrine : calcul des distances moyenne entre les atomes de la chaîne lourde D et de la chaîne légère M qui sont en contact étroit.
|
Code PDB : 2NBT Modifier les lignes suivantes du script "Contact.py" :
|
|
Résultats : distance minimale 4.63 / distance maximale : 201.62
|
Résultats : distance minimale 4.52 / distance maximale : 48.95
|
| Voir l'application en ligne. | |
Voir un développement sur l'implication du manteau de clathrine dans l'endocytose. |
Voir un développement sur la toxine de serpent de Bungarus multicinctus. |
|
Le programme MODELLER est un logiciel de modélisation de structures de protéines par homologie.
MODELLER effectue des tâches supplémentaires :
Format PIR/NBRF Format des fichiers de la base de données PIR ("Protein Information Resource"), émanation de NBRF ("National Biomedical Research Foundation").
Exemple >P1;CRAB_ANAPL ALPHA CRYSTALLIN B CHAIN MDITIHNPLI RRPLFSWLAP SRIFDQIFGE HLQESELLPA AQRK* |
a. Description de la procédure du programme MODELLER La recherche de séquences similaires dont les structures sont connues est effectuée avec la commande "profile. build ()" (contenue dans le script "build_profile.py") du programme MODELLER. Le script "build_profile. py" effectue les opérations suivantes : 1. Il initialise l'environnement de la modélisation en créant un objet "environ" (appelé "env"). Presque tous les scripts MODELLER nécessitent cette étape, car ce nouvel objet est nécessaire pour construire la plupart des autres objets utiles à la modélisation. 2. Il crée un objet "sequence_db" (appelé "sdb") utilisé pour contenir de grandes bases de données de séquences de protéines. 3. Il lit, dans la base de données "sdb", un fichier (au format texte) contenant des séquences non redondantes de la PDB. Les séquences sont aussi dans le fichier "pdb_95.pir" au format PIR. Chaque séquence de ce fichier est représentative d'un groupe de séquences de la PDB qui ont 95% ou plus d'identité de séquence et moins de 30 résidus ou moins de 30% de différence de longueur de séquence. 4a. Il écrit un fichier au format binaire contenant toutes les séquences lues à l'étape précédente. 4b. Il relit le fichier au format binaire pour une exécution plus rapide du script.
5a. Il crée un objet "alignment" ("aln"). 5b. Il lit la séquence cible TvLDH (dans l'exemple choisi) à partir du fichier "TvLDH.ali". 5c. Il convertit la séquence cible lue en un objet "profil " (appelé "prf"). Les profils contiennent des informations similaires à celles des alignements, mais ils sont plus compacts, plus complets et plus performants pour une recherche dans la base de données de séquences. 6. L'instruction "prf.build()" effectue la recherche dans "sdb" avec le profil "prf". Les correspondances qui sont trouvées sont ajoutées au profil. 7. L'instruction "prf.write()" écrit le nouveau profil contenant la séquence cible et ses homologues dans le fichier (au format texte) de sortie spécifié (le fichier "build_profile.prf"). 8. Conversion du nouveau profil en un nouvel alignement. 9. L'instruction "aln.write()" écrit le nouvel alignement contenant la séquence cible et ses homologues dans le fichier (au format PIR) de sortie spécifié (le fichier "build_profile.ali"). Récupérer le script "build_profile.py". Exécution du script Si le fichier est enregistré avec le nom "toto.py" et si la version de MODELLER est 9.19 (juillet 2017), la commande pour exécuter ce script est "mod9.19 toto.py".
Voir un descriptif très précis de toutes les étapes de MODELLER. |
b. Détail de la syntaxe de la commande profile.build() Cette commande analyse (de manière itérative) une base de données contenant un très grand nombre de séquences protéiques dans le but de créer un profil pour la séquence (ou l'alignement) que l'on veut modéliser. Cette commande calcule le score d'un alignement local (méthode de Smith-Waterman) entre la séquence à modéliser et chacune des séquences de la base de données. build( Les alignements avec des E-values inférieures à la valeur du paramètre "max_aln_evalue" sont ajoutés à l'alignement en cours. Un profil PSSM est générée à partir de l'alignement en cours : cette matrice est utilisée pour rechercher dans la base de données de séquences choisies. Cette procédure itérative est répétée :
Remarque : la recherche d'un repliement nécessite un drapeau "gaps_in_target=True". |
c. Bases de données liées à MODELLER ou utilisant ce programme α. Le serveur de prédiction de structures de protéines et le programme HHpred
HHpred :
β. La base de données de modèles de structures protéiques annotées ModBase ("Database of Comparative Protein Structure Models") Les modèles sont calculés par ModPipe, une suite logicielle ("pipeline") de modélisation automatisée qui repose principalement sur MODELLER pour :
ModBase contient plus de 5 millions de modèles fiables de domaines issus de presque 1,6 millions de séquences protéiques uniques. Seuls les modèles basés sur des alignements statistiquement significatifs et/ou des modèles dont le repliement est correct sont inclus. |
d. Exemples d'autres logiciels de modélisation structurale par homologie
|
| 12. Liens Internet et références bibliographiques | |
|
Atelier "Drug design" - Expasy Introduction to Biological Assemblies and the PDB Archive SWISS-MODEL Repository (SMR) : base de données de modèles de structures protéiques 3D annotées. Protein Family Models (HMMs) |
|
|
What are protein domains ? PSI-BLAST Tutorial : principe des profils / matrices PSSM. PSSMSearch : site web pour découvrir des motifs protéiques. |
|
|
Lee B. & Richards F.M. (1971) "The interpretation of protein structures : estimation of static accessibility" J. Mol. Biol. 55, 379 - 400 Kyte & Doolittle (1982) "A simple method for displaying the hydropathic character of a protein" J. Mol. Biol. 157, 105 - 132 Eisenberg et al. (1982) "The helical hydrophobic moment: a measure of the amphiphilicity of a helix" Nature 299, 371 - 374 Bailey & Elkan (1994) "Fitting a mixture model by expectation maximization to discover motifs in biopolymers", Proceedings of the Second International Conference on Intelligent Systems for Molecular Biology, pp. 28 - 36, AAAI Press, Menlo Park, California Berman et al. (2000) "The Protein Data Bank" Nucleic Acids Res. 28, 235 - 242 Ye & Godzik (2003) "Flexible structure alignment by chaining aligned fragment pairs allowing twists" Bioinformatics 19, ii246-ii255. |
----- |
|
Wu et al. (2003) "The Protein Information Resource" Nuc. Acids Res. 31, 345 - 347 Eddy S.R. (2004) "What is a hidden Markov model ?" Nat. Biotechnol. 22, 1315-1316 Zhang & Skolnick (2004) "Scoring function for automated assessment of protein structure template quality" Proteins 57, 702 - 710 Zhang & Skolnick (2005) "TM-align: a protein structure alignment algorithm based on the TM-score" Nucleic Acids Res. 33, 2302 - 2309 Eswar et al. (2006) "Comparative Protein Structure Modeling with MODELLER" Current Protocols in Bioinformatics, John Wiley & Sons, Inc., Supp. 15, 5.6.1-5.6.30 |
|
|
Schaeffer & Daggett (2011) "Protein folds and protein folding" Protein Eng. Des. Sel. 24, 11 - 19 Tien et al. (2013) "PeptideBuilder: A simple Python library to generate model peptides" PeerJ. 1, e80 Ali et al. (2014) "A review of methods available to estimate solvent-accessible surface areas of soluble proteins in the folded and unfolded states" Curr. Protein Pept. Sci. 15, 456 - 476 Webb & Sali (2016) "Comparative protein structure modeling using MODELLER" Curr. Protoc. Bioinformatics Chapter 5, unit 5.6 Finn et al. (2016) "The Pfam protein families database: towards a more sustainable future" Nuc. Acids Res. Database Issue 44, D279 - D285 |
|
|
Janson et al. (2017) "PyMod 2.0: improvements in protein sequence-structure analysis and homology modeling within PyMOL" Bioinformatics 33, 444 - 446 Jubb et al. (2017) "Arpeggio: a web server for calculating and visualising interatomic interactions in protein structures" J. Mol. Biol. 429, 365 - 371 Burley et al. (2018) "RCSB Protein Data Bank: Sustaining a living digital data resource that enables breakthroughs in scientific research and biomedical education" Protein Sci. 27, 316 - 330 |
|
|
Mitchell et al. (2019) "InterPro in 2019: improving coverage, classification and access to protein sequence annotations" Nucleic Acids Res. 47 (Database issue), D351 - D360 Steinegger et al. (2019) "HH-suite3 for fast remote homology detection and deep protein annotation" BMC Bioinformatics 20, 473 Wiltgen M. (2019) "Algorithms for Structure Comparison and Analysis: Homology Modelling of Proteins" in "Encyclopedia of Bioinformatics and Computational Biology" |
|
|
Jumper et al. (2021) "Highly accurate protein structure prediction with AlphaFold" Nature 596, 583 - 589 Wang et al. (2021) « Protein domain identification methods and online resources » Comput. Struct. Biotechnol. J. 19, 1145 - 1153 Bileschi et al. (2022) "Using deep learning to annotate the protein universe" Nat. Biotechnol. 40, 932 - 937 Mohammadi et al. (2022) "PSSMCOOL: a comprehensive R package for generating evolutionary-based descriptors of protein sequences from PSSM profiles" Biol. Methods Protoc. 7, bpac008 Liu et al. (2024) "PLMSearch: Protein language model powers accurate and fast sequence search for remote homology" Nat. Commun. 15, 2775 |
|